Самоорганизующиеся карты (СОМ) — это тип искусственной нейронной сети, используемой для нед supervised learning задач, таких как кластеризация. Учитывая набор данных, модель учится делить данные на кластеры. СОМ проецируют сложные многомерные данные на более низкоразмерную (обычно 2-мерную) сетку. Это облегчает визуализацию. Таким образом, СОМ также используются для визуализации высокоразмерных данных.
В этом руководстве мы исследуем основные концепции СОМ, их процесс обучения и типичные случаи использования. Мы объясняем, как реализовать СОМ на Python с использованием библиотеки MiniSom и как визуально представить результаты. Наконец, мы обсуждаем важные гиперпараметры в обучении моделей СОМ и как их тонко настраивать.
Понимание самоорганизующихся карт (СОМ)
В этом разделе мы представляем основные концепции самоорганизующихся карт (СОК), их процесс обучения и области применения.
Основные концепции СОК
Самоорганизующиеся карты включают в себя набор концепций, которые важно понимать.
Нейроны
СОК по сути представляет собой сетку нейронов. В процессе обучения нейрон, весовой вектор которого ближе всего к входной точке данных, корректирует свои веса и веса соседей, чтобы еще более точно соответствовать входной точке данных. За множество итераций группы соседних нейронов отображаются на связанные точки данных. Это приводит к кластеризации входного набора данных.
Структура сетки
Нейроны в SOM организованы в виде сетки. Соседние нейроны соответствуют похожим точкам данных. Для больших наборов данных нужна большая сетка. Обычно эта сетка является двумерной. Структура сетки служит низкоразмерным пространством, в которое отображаются высокоразмерные данные. Сетка помогает визуализировать шаблоны данных и кластеры.
Отображение точек данных
Каждая точка данных сравнивается со всеми нейронами с использованием метрики расстояния. Нейрон, чей вектор весов ближе всего к точке входных данных, является Лучшим Соответствующим Элементом (BMU) для этой точки данных.
Как только выбрана лучшая нейронная единица (BMU), обновляются веса BMU и соседних нейронов. Это обновление приближает BMU и соседние нейроны к входной точке данных. Это отображение сохраняет топологию данных, обеспечивая, чтобы похожие точки данных отображались на соседние нейроны.
Процесс обучения SOM
Обучение SOM считается неконтролируемым, поскольку не основано на размеченных наборах данных. Цель обучения SOM заключается в итеративной настройке векторных весов нейронов так, чтобы похожие точки данных отображались на соседние нейроны.
Конкурентное обучение
SOM используют конкурентное обучение (вместо градиентного спуска и обратного распространения ошибки). Нейроны соревнуются за то, чтобы стать Лучшим Соответствующим Элементом (BMU) для каждой точки входных данных. Нейрон, ближайший к точке данных, определяется как ее BMU. BMU и его соседние нейроны обновляются, чтобы еще больше уменьшить их расстояние до точки данных. Соседние нейроны отображаются на связанные точки данных. Это приводит к специализации среди нейронов и кластеризации входных данных.
Функции расстояния
SOM используют функцию расстояния для измерения расстояния между нейронами и точками данных. Это расстояние используется для определения BMU каждой точки данных. MiniSom имеет четыре функции расстояния на выбор:
- Евклидово расстояние (это выбор по умолчанию)
- Косинусное расстояние
- Манхэттенское расстояние
- Расстояние Чебышева
Функция соседства
После определения BMU (лучшего совпадающего нейрона) для точки данных, BMU и его соседние нейроны обновляются в направлении этой точки данных. Функция соседства обеспечивает сохранение топологических взаимосвязей входных данных в SOM. Функция соседства определяет:
- Какие нейроны считаются соседями BMU
- Степень, в которой обновляются соседние нейроны. В общем, нейроны, находящиеся ближе к BMU, получают большее изменение, чем те, которые находятся дальше
MiniSom поставляется с тремя функциями соседства:
- Гауссовская (это значение по умолчанию).
- Пузырь
- Мексиканская шляпа
- Треугольник
Итеративный процесс
Процесс обучения в SOM происходит за несколько итераций. На каждой итерации SOM обрабатывает множество входных данных. Мы описываем процесс обучения в следующих шагах:
- Когда начинается обучение, веса всех нейронов (случайно) инициализируются.
- Учитывая входную точку данных, каждый нейрон вычисляет свое расстояние от входа.
- Нейрон с наименьшим расстоянием объявляется BMU.
- Веса BMU и его соседей корректируются, чтобы приблизиться к входному вектору. Степень этой корректировки определяется:
- Функцией соседства: нейроны, находящиеся дальше от BMU, обновляются меньше, чем те, что ближе к BMU. Это сохраняет топологическую структуру данных.
- Скорость обучения: Более высокая скорость обучения приводит к большим обновлениям.
Эти шаги повторяются на протяжении многих итераций, проходя через множество входных данных. Веса постепенно обновляются, чтобы карта самоорганизовывалась и захватывала структуру данных.
Скорость обучения и радиус соседства обычно уменьшаются со временем, позволяя SOM постепенно настраивать веса. Позже в учебнике мы реализуем шаги итеративного процесса обучения с использованием кода Python.
Типичные случаи использования SOM
Учитывая их уникальную архитектуру, SOM имеют несколько типичных приложений в машинном обучении, анализе и визуализации.
Кластеризация
SOM используются для группировки многомерных данных в кластеры похожих точек данных. Это помогает выявить внутренние структуры в данных. Каждый нейрон соответствует тесно связанным точкам данных, и похожие точки данных отображаются на один и тот же или соседние нейроны, образуя тем самым отчетливые кластеры.
Например, в маркетинге SOM могут группировать клиентов на основе покупательского поведения. Это позволит бренду адаптировать маркетинговые стратегии к различным сегментам клиентов (кластерам).
Снижение размерности
SOM могут отображать многомерные данные с множеством векторных признаков на сетку с меньшим количеством размерностей (обычно, двумерную сетку). Поскольку это отображение сохраняет отношения между точками данных, сниженная размерность облегчает визуализацию сложных наборов данных без значительной потери информации.
В дополнение к упрощению задач визуализации и анализа, СОМы также могут уменьшать размерность данных перед применением других алгоритмов машинного обучения. СОМы также используются в обработке изображений для уменьшения числа признаков путем кластеризации пикселей или областей с похожими характеристиками, что делает задачи распознавания и классификации изображений более эффективными.
Обнаружение аномалий
В процессе обучения большинство точек данных отображаются на определенные нейроны. Аномалии (точки данных, которые плохо вписываются в какой-либо кластер) обычно отображаются на удаленных или менее населенных нейронах. Эти выбросы плохо вписываются в изученные кластеры и отображаются на нейронах, находящихся далеко от BMU нормальных точек данных.
Например, отображение финансовых транзакций на сетке СОМ позволяет выявлять необычные действия (выбросы), которые могут указывать на потенциально мошенническую деятельность.
Визуализация данных
Визуализация данных является одним из самых распространенных вариантов использования SOM. Высокоразмерные данные сложно визуализировать. SOM упрощают визуализацию, уменьшая размерность данных и проецируя их на двумерную сетку. Это также может помочь обнаружить кластеры в данных и аномальные точки данных, которые было бы сложно выявить в исходных данных.
Например, визуализация данных упрощает анализ высокоразмерных данных о населении. Отображение данных на 2D сетке помогает выявить сходства и различия между различными группами населения.
После обсуждения основных концепций и вариантов использования SOM мы покажем, как их реализовать с помощью пакета Python MiniSom в следующих разделах.
Вы можете получить доступ к полному коду и запустить его на этом блокноте DataLab.
Настройка окружения для SOM
Перед созданием SOM нам необходимо подготовить окружение с необходимыми пакетами.
Установка библиотек Python
Нам нужны следующие пакеты:
- MiniSom — это инструмент на основе NumPy для создания и обучения SOM.
- NumPy используется для доступа к математическим функциям, таким как разделение массивов, получение уникальных значений и т.д.
matplotlibиспользуется для построения различных графиков и диаграмм для визуализации данных.- Пакет
datasetsизsklearnиспользуется для импорта наборов данных, к которым будет применяться SOM. - Пакет
MinMaxScalerизsklearnнормализует набор данных.
Следующий фрагмент кода импортирует эти пакеты:
from minisom import MiniSom import numpy as np import matplotlib.pyplot as plt from sklearn import datasets from sklearn.preprocessing import MinMaxScaler
Подготовка набора данных
В этом учебном пособии мы используем MiniSom для построения SOM, а затем обучаем его на каноническом наборе данных IRIS. Этот набор данных состоит из 3 классов ирисов. Каждый класс содержит 50 экземпляров. Чтобы подготовить данные, мы выполняем следующие шаги:
- Импортируем набор данных Iris из
sklearn, - Извлекаем векторы данных и целевые скаляры.
- Нормализуйте векторы данных. В этом руководстве мы используем MinMaxScaler из scikit-learn.
- Объявите набор меток для каждого из трех классов растений Ирис.
Следующий код реализует эти шаги:
dataset_iris = datasets.load_iris() data_iris = dataset_iris.data target_iris = dataset_iris.target data_iris_normalized = MinMaxScaler().fit_transform(data_iris) labels_iris = {1:'1', 2:'2', 3:'3'} data = data_iris_normalized target = target_iris
Реализация самоорганизующихся карт (SOM) на Python
Чтобы реализовать SOM на Python, мы определяем и инициализируем сетку перед обучением ее на наборе данных. Затем мы можем визуализировать обученные нейроны и сгруппированный набор данных.
Определение сетки SOM
Как уже объяснялось ранее, SOM представляет собой сетку нейронов. С помощью MiniSom мы можем создать двумерные сетки. Размеры сетки по осям X и Y определяются количеством нейронов вдоль каждой оси. Чтобы определить сетку SOM, нам также нужно указать:
- Размеры сетки по осям X и Y
- Количество входных переменных – это количество строк данных.
Объявите эти параметры как константы Python:
SOM_X_AXIS_NODES = 8 SOM_Y_AXIS_NODES = 8 SOM_N_VARIABLES = data.shape[1]
Пример кода ниже иллюстрирует, как объявить сетку с использованием MiniSom:
som = MiniSom(SOM_X_AXIS_NODES, SOM_Y_AXIS_NODES, SOM_N_VARIABLES)
Первые два параметра – это количество нейронов вдоль осей X и Y, а третий параметр – это количество переменных.
Мы объявляем другие параметры и гиперпараметры при создании сетки SOM. Мы объясним их позже в учебнике. Пока объявите эти параметры, как показано ниже:
ALPHA = 0.5 DECAY_FUNC = 'linear_decay_to_zero' SIGMA0 = 1.5 SIGMA_DECAY_FUNC = 'linear_decay_to_one' NEIGHBORHOOD_FUNC = 'triangle' DISTANCE_FUNC = 'euclidean' TOPOLOGY = 'rectangular' RANDOM_SEED = 123
Создайте SOM, используя эти параметры:
som = MiniSom( SOM_X_AXIS_NODES, SOM_Y_AXIS_NODES, SOM_N_VARIABLES, sigma=SIGMA0, learning_rate=ALPHA, neighborhood_function=NEIGHBORHOOD_FUNC, activation_distance=DISTANCE_FUNC, topology=TOPOLOGY, sigma_decay_function = SIGMA_DECAY_FUNC, decay_function = DECAY_FUNC, random_seed=RANDOM_SEED, )
Инициализация нейронов
Вышеуказанная команда создает SOM со случайными весами для всех нейронов. Инициализация нейронов с весами, взятыми из данных (вместо случайных чисел), может сделать процесс обучения более эффективным.
При использовании MiniSom для создания самоорганизующейся карты (SOM) есть два способа инициализации весов нейронов на основе данных:
- Случайная инициализация: Начальные веса нейронов случайным образом выбираются из входных данных. Мы делаем это, применяя функцию
.random_weights_init()к СОМ. - Инициализация PCA: Инициализация с помощью метода главных компонент (PCA) использует главные компоненты входных данных для инициализации весов. Начальные веса нейронов охватывают первые две главные компоненты. Это часто приводит к более быстрой сходимости.
В этом руководстве мы используем инициализацию PCA. Чтобы применить инициализацию PCA к весам SOM, используйте функцию .pca_weights_init() как показано ниже:
som.pca_weights_init(data)
Обучение SOM
Процесс обучения обновляет веса SOM, чтобы минимизировать расстояние между нейронами и точками данных.
Ниже мы объясняем итеративный процесс обучения:
- Инициализация: Векторы весов всех нейронов инициализируются, как правило, случайными значениями. Также возможно инициализировать веса, выбирая из распределения входных данных.
- Выбор входных данных: Входной вектор (случайным образом) выбирается из обучающего набора данных.
- Идентификация BMU: Нейрон с вектором весов, наиболее близким к входному вектору, идентифицируется как BMU.
- Обновление соседей: BMU и его соседние нейроны обновляют свои векторные весы. Скорость обучения и функция соседства определяют, какие нейроны обновляются и на сколько. На шаге итерации t, учитывая входной вектор x, вектор весов нейрона i равен wi, скорость обучения (t), и функция соседства hbi (эта функция количественно оценивает степень обновления для нейрона i с учетом нейрона BMU b), формула обновления веса для нейрона i выражается как:

- Скорость уменьшения скорости обучения и радиуса соседства: Как скорость обучения, так и радиус соседства уменьшаются со временем. На более ранних итерациях процесс обучения вносит большие изменения в более широком окружении. Поздние итерации помогают уточнить веса, внося меньшие изменения в веса соседних нейронов. Это позволяет карте стабилизироваться и сходиться.
Чтобы обучить SOM, мы представляем модели входные данные. Мы можем выбрать один из двух подходов для этого:
- Выбирать образцы случайным образом из входных данных. Функция
.train_random()реализует эту технику. - Последовательно обрабатывать векторы во входных данных. Это делается с помощью функции
.train_batch()функция.
Эти функции принимают входные данные и количество итераций в качестве параметров. В этом руководстве мы используем функцию .train_random() функция. Объявите количество итераций как константу и передайте его в функцию обучения:
N_ITERATIONS = 5000 som.train_random(data, N_ITERATIONS, verbose=True)
После выполнения скрипта и завершения обучения отображается сообщение с ошибкой квантизации:
quantization error: 0.05357240680504421
Ошибка квантования указывает на количество информации, потерянной при квантовании (уменьшении размерности) данных в SOM. Большая ошибка квантования указывает на большее расстояние между нейронами и точками данных. Это также означает, что кластеризация менее надежна.
Визуализация нейронов SOM
Теперь у нас есть обученная модель SOM. Чтобы визуализировать ее, мы используем карту расстояний (также известную как U-матрица). Карта расстояний отображает нейроны SOM в виде сетки ячеек. Цвет каждой ячейки представляет собой расстояние до соседних нейронов.
Карта расстояний представляет собой сетку с такими же размерами, как и SOM. Каждая ячейка на карте расстояний является нормализованной суммой (евклидовых) расстояний между нейроном и его соседями.
Получите доступ к карте расстояний SOM с помощью функции .distance_map(). Чтобы сгенерировать U-матрицу, следуем этим шагам:
- Используйте
pyplotдля создания фигуры с теми же размерами, что и SOM. В этом примере размеры составляют 8×8. - Постройте карту расстояний, используя matplotlib и функцию
.pcolor(). В этом примере мы используемgist_yargв качестве цветовой схемы. - Отобразите
colorbar, индекс, сопоставляющий различные цвета с различными скалярными значениями. В данном случае, поскольку расстояния нормализованы, скалярные значения расстояний варьируются от 0 до 1.
Код ниже реализует эти шаги:
# создайте сетку plt.figure(figsize=(8, 8)) # постройте карту расстояний plt.pcolor(som.distance_map().T, cmap='gist_yarg') # покажите цветовую шкалу plt.colorbar() plt.show()
В этом примере U-матрица использует монотонную цветовую схему. Это можно понять, следуя этим рекомендациям:
- Светлые оттенки представляют нейроны, расположенные близко друг к другу, а темные оттенки обозначают нейроны, находящиеся дальше от других.
- Группы более светлых оттенков могут быть интерпретированы как кластеры. Темные узлы между кластерами могут быть интерпретированы как границы между кластерами.
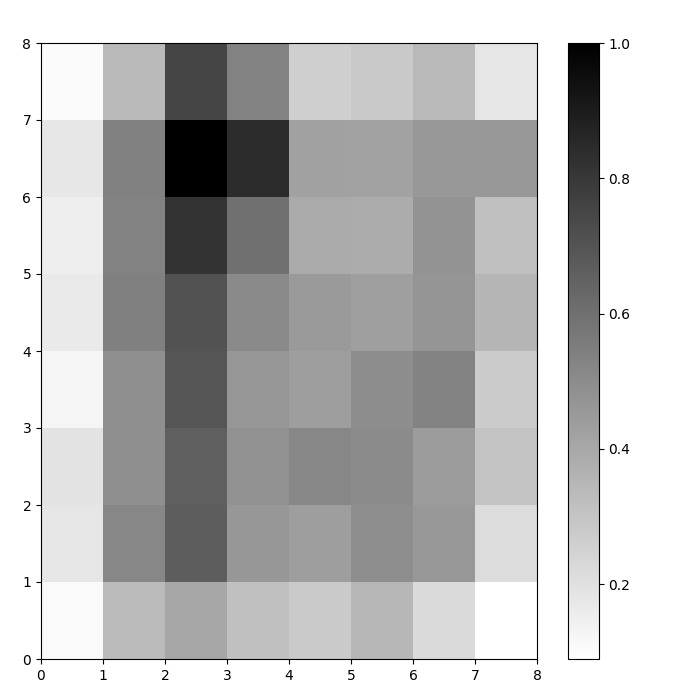
Рисунок 1: U-матрица SOM, обученная на наборе данных Iris (изображение автора)
Оценка результатов кластеризации SOM
Предыдущий рисунок графически иллюстрировал нейроны SOM. В этом разделе мы покажем, как визуализировать, как SOM кластеризовал данные.
Идентификация кластеров
Мы накладываем маркеры на вышеуказанную U-матрицу, чтобы обозначить, какой класс растения Iris представляет каждая ячейка (нейрон). Для этого:
- Как и раньше, создайте фигуру 8×8 с использованием
pyplot, постройте карту расстояний и отобразите цветовую шкалу. - Укажите массив из трех маркерoв matplotlib, по одному для каждого класса растения Iris.
- Укажите массив из трех цветовых кодов matplotlib, по одному для каждого класса растения Iris.
- Итеративно изображайте выигравший нейрон для каждой точки данных:
- Определите (координаты) выигравшего нейрона для каждой точки данных, используя функцию
.winner(). - Постройте положение каждого выигравшего нейрона в центре каждой ячейки на сетке.
w[0]иw[1]дают X и Y координаты нейрона соответственно. К каждой координате добавляется значение 0.5, чтобы отобразить его в центре ячейки.
Ниже приведён код, который показывает, как это сделать:
# постройте карту расстояний plt.figure(figsize=(8, 8)) plt.pcolor(som.distance_map().T, cmap='gist_yarg') plt.colorbar() # создайте маркеры и цвета для каждого класса markers = ['o', 'x', '^'] colors = ['C0', 'C1', 'C2'] # постройте выигравший нейрон для каждой точки данных for count, datapoint in enumerate(data): # получите победителя w = som.winner(datapoint) # разместите маркер на выигрышной позиции для образца точки данных plt.plot(w[0]+.5, w[1]+.5, markers[target[count]-1], markerfacecolor='None', markeredgecolor=colors[target[count]-1], markersize=12, markeredgewidth=2) plt.show()
Полученное изображение показано ниже:
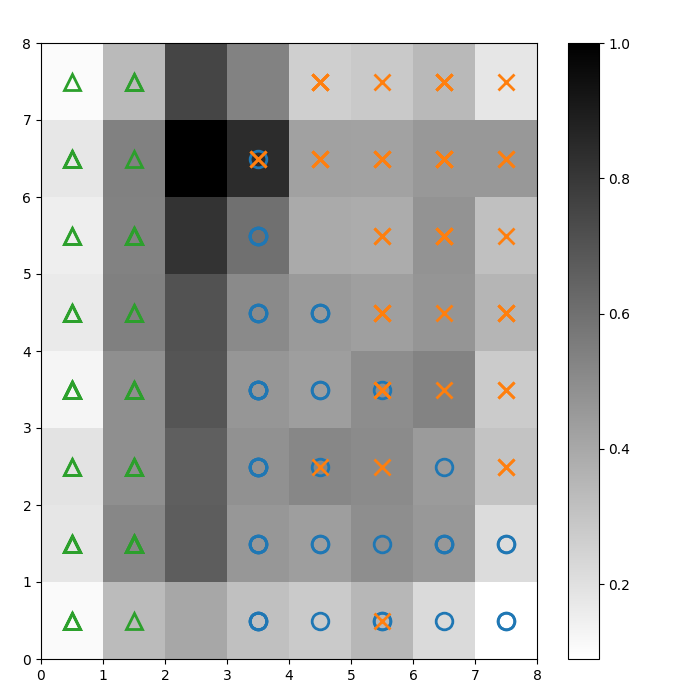
Рисунок 2: U-матрица с наложенными маркерами классов (изображение автора)
Основываясь на документации датасета Iris, “один класс линейно отделим от других 2; последние не являются линейно отделимыми друг от друга”. В U-матрице выше эти три класса представлены тремя маркерами – треугольником, кругом и крестом.
Обратите внимание, что между синими кругами и оранжевыми крестами нет четкой границы. Кроме того, два класса накладываются на один и тот же нейрон во многих ячейках. Это означает, что нейрон находится на равном расстоянии от обоих классов.
Визуализация результата кластеризации
СОМ — это модель кластеризации. Похожие данные сопоставляются с одним и тем же нейроном. Данные одной и той же категории сопоставляются с кластером соседних нейронов. Мы отображаем все данные на сетке СОМ, чтобы лучше изучить поведение кластеризации.
Следующие шаги описывают, как создать этот разбросанный график:
- Получите X и Y координаты выигравшего нейрона для каждой точки данных.
- Постройте карту расстояний, как мы сделали для Рисунок 1.
- Используйте
plt.scatter(), чтобы построить точечную диаграмму всех выигравших нейронов для каждой точки данных. Добавьте случайное смещение к каждой точке, чтобы избежать перекрытия между точками данных в одной ячейке.
Мы реализуем эти шаги в коде ниже:
# получаем X и Y координаты выигравшего нейрона для каждой точки данныхw_x, w_y = zip(*[som.winner(d) for d in data]) w_x = np.array(w_x) w_y = np.array(w_y) # строим карту расстояний plt.figure(figsize=(8, 8)) plt.pcolor(som.distance_map().T, cmap='gist_yarg', alpha=.2) plt.colorbar() # создаем точечную диаграмму всех выигравших нейронов для каждой точки данных # добавляем случайное смещение к каждой точке, чтобы избежать перекрытий for c in np.unique(target): idx_target = target==c plt.scatter(w_x[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8, w_y[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8, s=50, c=colors[c-1], label=labels_iris[c+1] ) plt.legend(loc='upper right') plt.grid() plt.show()
Следующий график показывает результирующую точечную диаграмму:
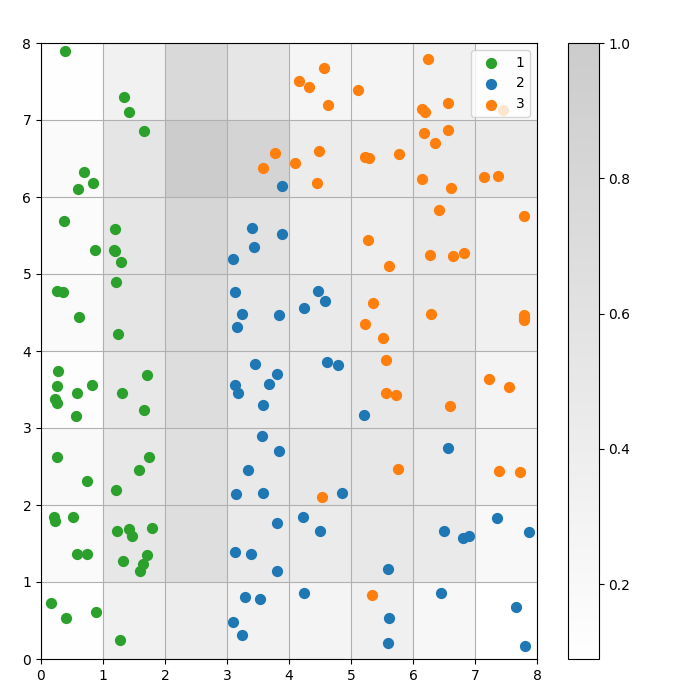 Рисунок 3: Диаграмма рассеяния точек данных в ячейках (изображение автора)
Рисунок 3: Диаграмма рассеяния точек данных в ячейках (изображение автора)
На вышеуказанной диаграмме рассеяния обратите внимание на:
- Некоторые ячейки содержат как синие, так и оранжевые точки.
- Зеленые точки явно отделены от остальных данных, но синие и оранжевые точки не отделены четко.
- Вышеуказанные наблюдения соответствуют тому факту, что только один из трех кластеров в наборе данных Iris имеет четкую границу.
- В Рисунке 1, темные узлы между кластерами (которые можно интерпретировать как границы между кластерами) совпадают с пустыми ячейками на диаграмме рассеяния.
Вы можете получить доступ и запустить полный код в этом блокноте DataLab.
Настройка модели SOM
В предыдущих разделах показано, как создать и обучить модель SOM, а также как визуально изучить результаты. В этом разделе мы обсуждаем, как настроить производительность моделей SOM.
Ключевые гиперпараметры для настройки
Как и в случае с любой моделью машинного обучения, гиперпараметры значительно влияют на производительность модели.
Некоторые из гиперпараметров, важных для обучения SOM, включают:
- Размер сетки определяет размер карты. Количество нейронов на карте с размером сетки AxB составляет A*B.
- Скорость обучения определяет, насколько изменяются веса на каждой итерации. Мы устанавливаем начальную скорость обучения, и она уменьшается со временем в соответствии с функцией убывания.
- Функция убывания определяет степень, на которую скорость обучения уменьшается на каждой последующей итерации.
- Функциясоседства — это математическая функция, которая определяет, какие нейроны будут считаться соседями БМУ.
- Стандартноеотклонение определяет разброс функции соседства. Например, гауссовская функция соседства с высоким стандартным отклонением будет иметь большее соседство, чем та же функция с меньшим стандартным отклонением. Мы устанавливаем начальное стандартное отклонение, которое уменьшается со временем в соответствии с функцией затухания сигмы.
- Функция сигма-распад контролирует, насколько стандартное отклонение уменьшается на каждой последующей итерации.
- Количество итераций обучения определяет, сколько раз обновляются веса. На каждой итерации обучения веса нейронов обновляются один раз.
- Функция расстояния — это математическая функция, которая вычисляет расстояние между нейронами и данными.
- Топология определяет структуру сетки. Нейроны в сетке могут быть расположены в прямоугольном или шестиугольном паттерне.
В следующем разделе мы обсудим рекомендации по установке значений этих гиперпараметров.
Влияние настройки гиперпараметров
Значения гиперпараметров должны определяться на основе модели и набора данных. В какой-то степени определение этих значений является процессом проб и ошибок. В этом разделе мы даем рекомендации по настройке каждого гиперпараметра. Рядом с каждым гиперпараметром мы указываем (в скобках) соответствующие константы Python, используемые в примере кода.
- Размер сетки (
SOM_X_AXIS_NODESиSOM_X_AXIS_NODES): Размер сетки зависит от размера набора данных. Правило заключается в том, что для набора данных размером N, сетка должна содержать примерно 5*sqrt(N) нейронов. Например, если набор данных содержит 150 образцов, сетка должна содержать 5*sqrt(150) = примерно 61 нейрон. В этом учебном пособии набор данных Iris содержит 150 строк, и мы используем сетку 8×8. - Начальная скорость обучения (
ALPHA): Более высокая скорость ускоряет сходимость, в то время как более низкие скорости используются для более тонкой настройки после ранних итераций. Начальная скорость обучения должна быть достаточно большой, чтобы обеспечить быструю адаптацию, но не настолько большой, чтобы превышать оптимальные значения весов. В этой статье начальная скорость обучения составляет 0.5. - Начальное стандартное отклонение (
SIGMA0): Оно определяет начальный размер или разброс окрестности. Более крупное значение учитывает более глобальные паттерны. В этом примере мы используем начальное стандартное отклонение 1.5. - Для коэффициента распада (
DECAY_FUNC) и коэффициента сигма распада (SIGMA_DECAY_FUNC), мы можем выбрать один из трех типов функций распада: - Обратное затухание: Эта функция подходит, если данные имеют как глобальные, так и локальные паттерны. В таких случаях нам нужна более длительная фаза широкого обучения, прежде чем сосредоточиться на локальных паттернах.
- Линейное затухание: Это хорошо подходит для наборов данных, где мы хотим стабильный и равномерный размер соседства или снижение скорости обучения. Это полезно, если данным не нужно много тонкой настройки.
- Ассимптотическое затухание: Эта функция полезна, если данные сложные и многомерные. В таких случаях лучше потратить больше времени на глобальное исследование, прежде чем постепенно переходить к более тонким деталям.
- Функция соседства (
NEIGHBORHOOD_FUNC): По умолчанию используется функция Гаусса. Также применяются другие функции, как объяснено ниже. - Гауссова (по умолчанию): Это кривая в форме колокола. Степень обновления нейрона плавно уменьшается по мере увеличения расстояния от выигравшего нейрона. Она обеспечивает плавный и непрерывный переход и сохраняет топологию данных. Подходит для большинства общих целей благодаря своей стабильной и предсказуемой работе.
- Пузыри: Эта функция создает фиксированное по ширине соседство. Все нейроны внутри этого соседства обновляются одинаково, а нейроны за его пределами не обновляются (для данной точки данных). Это вычислительно дешевле и проще в реализации. Это полезно для маленьких карт, где резкие границы соседства не компрометируют эффективную кластеризацию.
- Мексиканская шляпа: У нее есть центральная положительная область, окруженная отрицательной областью. Нейроны, близкие к BMU, обновляются, чтобы приблизиться к точке данных, а нейроны, находящиеся дальше, обновляются, чтобы удалиться от точки данных. Эта техника усиливает контраст и уточняет характеристики на карте. Так как она подчеркивает отдельные кластеры, она эффективна в задачах распознавания образов, где требуется четкое разделение кластеров.
- Треугольник: Эта функция определяет размер окрестности как треугольник, при этом BMU имеет наибольшее влияние. Он уменьшается линейно с расстоянием от BMU. Используется для кластеризации данных с постепенными переходами между кластерами или признаками, такими как изображения, речь или временные ряды, где соседние точки данных ожидаются как имеющие схожие характеристики.
- Функция расстояния (
DISTANCE_FUNC): Для измерения расстояния между нейронами и точками данных мы можем выбрать из 4 методов: - Евклидово расстояние (выбор по умолчанию): Полезно, когда данные непрерывные, и мы хотим измерить расстояние по прямой. Оно подходит для большинства общих задач, особенно когда точки данных равномерно распределены и пространственно связаны.
- Косинусное расстояние: Хороший выбор для текста или разреженных данных высокой размерности, где угол между векторами более важен, чем величина. Оно полезно для сравнения направленности в данных.
- Манхэттенское расстояние: Идеально подходит, когда точки данных находятся на сетке или решетке (например, городские кварталы). Оно менее чувствительно к выбросам, чем Евклидово расстояние.
- Расстояние Чебышева: Подходит для ситуаций, когда движение может происходить в любом направлении (например, расстояния на шахматной доске). Полезно для дискретных пространств, где мы хотим приоритизировать максимальное различие по осям.
- Топология (
TOPOLOGY): В сетке нейроны могут быть расположены в гексагональной или прямоугольной структуре: - Прямоугольный (по умолчанию): каждый нейрон имеет 4 непосредственных соседа. Это правильный выбор, когда данные не имеют четкой пространственной связи. Это также вычислительно проще.
- Шестиугольный: каждый нейрон имеет 6 соседей. Это предпочитаемый вариант, если данные имеют пространственные связи, которые лучше представлены с помощью шестиугольной сетки. Это относится к круговым или угловым распределениям данных.
- Количество итераций обучения (
N_ITERATIONS): В принципе, более длительное время обучения приводит к снижению ошибок и лучшему соответствию весов входным данным. Однако производительность модели увеличивается асимптотически с количеством итераций. Таким образом, после определенного количества итераций увеличение производительности от последующих взаимодействий становится лишь незначительным. Определение правильного количества итераций требует некоторого эксперимента. В этом уроке мы обучаем модель в течение 5000 итераций.
Чтобы определить правильную конфигурацию гиперпараметров, мы рекомендуем экспериментировать с различными вариантами на меньшем подмножестве данных.
Заключение
Самоорганизующиеся карты являются надежным инструментом для обучения без учителя. Они используются для кластеризации, уменьшения размерности, обнаружения аномалий и визуализации данных. Поскольку они сохраняют топологические свойства многомерных данных и представляют их на сетке с меньшей размерностью, SOM облегчают визуализацию и интерпретацию сложных наборов данных.
В этом уроке обсуждаются основные принципы SOM и показывается, как реализовать SOM с использованием библиотеки MiniSom на Python. Также демонстрируется, как визуально проанализировать результаты и объясняются важные гиперпараметры, используемые для обучения SOM и настройки их производительности.
Source:
https://www.datacamp.com/tutorial/self-organizing-maps













