Mapas auto-organizáveis (SOMs) são um tipo de rede neural artificial usada para tarefas de aprendizado não supervisionado, como agrupamento. Dado um conjunto de pontos de dados, o modelo aprende a dividir os dados em clusters. Os SOMs projetam dados complexos em múltiplas dimensões em uma grade de menor dimensão (tipicamente, 2 dimensões). Isso facilita uma visualização mais fácil. Portanto, os SOMs também são usados para visualizar dados de alta dimensão.
Neste tutorial, exploramos os conceitos fundamentais dos SOMs, seu processo de aprendizado e seus casos de uso típicos. Explicamos como implementar um SOM em Python usando a biblioteca MiniSom e como representar os resultados visualmente. Por fim, discutimos hiperparâmetros importantes no treinamento de modelos SOM e como ajustá-los.
Entendendo Mapas Auto-Organizáveis (SOM)
Nesta seção, apresentamos os conceitos principais dos MAPs auto-organizáveis, seu processo de aprendizagem e seus casos de uso.
Conceitos principais dos MAPs auto-organizáveis
Os mapas auto-organizáveis envolvem um conjunto de conceitos que são importantes para entender.
Neurônios
Um MAP é essencialmente uma grade de neurônios. Durante o treinamento, o neurônio cujo vetor de peso está mais próximo de um ponto de dados de entrada ajusta seus pesos e os pesos dos vizinhos para se igualar ainda mais ao ponto de dados de entrada. Ao longo de muitas iterações, grupos de neurônios vizinhos mapeiam para pontos de dados relacionados. Isso resulta na agrupamento do conjunto de dados de entrada.
Estrutura da grade
Os neurônios em um SOM são organizados como uma grade. Neurônios vizinhos mapeiam para pontos de dados semelhantes. Conjuntos de dados maiores precisam de uma grade maior. Normalmente, essa grade é bidimensional. A estrutura da grade serve como o espaço de baixa dimensão para o qual os dados de alta dimensão são mapeados. A grade ajuda a visualizar padrões e clusters de dados.
Mapeando pontos de dados
Cada ponto de dado é comparado a todos os neurônios usando uma métrica de distância. O neurônio cujo vetor de peso é mais próximo do ponto de dado de entrada é a Unidade de Melhor Correspondência (BMU) para aquele ponto de dado.
Uma vez que a BMU é identificada, os pesos da BMU e de seus neurônios vizinhos são atualizados. Essa atualização aproxima ainda mais a BMU e os neurônios vizinhos do ponto de dados de entrada. Esse mapeamento preserva a topologia dos dados, garantindo que pontos de dados semelhantes sejam mapeados para neurônios próximos.
Processo de aprendizado das SOMs
O treinamento de SOM é considerado não supervisionado porque não é baseado em conjuntos de dados rotulados. O objetivo do treinamento das SOMs é ajustar iterativamente os vetores de peso dos neurônios para que pontos de dados semelhantes sejam mapeados para neurônios próximos.
Aprendizado competitivo
SOMs usam aprendizado competitivo (em vez de descida de gradiente e retropropagação). Os neurônios competem para se tornarem a Unidade de Melhor Correspondência (BMU) para cada ponto de dados de entrada. O neurônio mais próximo de um ponto de dados é determinado como seu BMU. O BMU e seus neurônios vizinhos são atualizados para diminuir ainda mais a distância em relação ao ponto de dados. Os neurônios vizinhos se mapeiam para pontos de dados relacionados. Isso leva à especialização entre os neurônios e ao agrupamento dos pontos de dados de entrada.
Funções de distância
SOMs usam uma função de distância para medir a distância entre neurônios e pontos de dados. Essa distância é usada para determinar o BMU de cada ponto de dados. O MiniSom tem quatro funções de distância para escolher:
- distância euclidiana (esta é a escolha padrão)
- Distância cosseno
- Distância Manhattan
- Distância de Chebyshev
Função de vizinhança
Após identificar o BMU de um ponto de dados, o BMU e seus neurônios vizinhos são atualizados na direção desse ponto de dados. A função de vizinhança garante que o SOM mantenha as relações topológicas dos dados de entrada. A função de vizinhança decide:
- Quais neurônios são considerados vizinhos do BMU
- A medida em que os neurônios vizinhos são atualizados. Em geral, neurônios mais próximos do BMU recebem um ajuste maior do que aqueles mais distantes
MiniSom vem com três funções de vizinhança:
- Gaussiana (esta é a padrão).
- Bolha
- Chapéu mexicano
- Triângulo
Processo iterativo
O processo de aprendizado em SOMs acontece ao longo de várias iterações. Em cada iteração, o SOM processa muitos pontos de dados de entrada. Esboçamos o processo de aprendizado nos seguintes passos:
- Quando o treinamento começa, os pesos de todos os neurônios são (aleatoriamente) inicializados.
- Dado um ponto de dados de entrada, cada neurônio calcula sua distância em relação à entrada.
- O neurônio com a menor distância é declarado o BMU.
- Os pesos do BMU e de seus vizinhos são ajustados para se aproximar do vetor de entrada. A extensão desse ajuste é decidida por:
- Uma função de vizinhança: Neurônios mais distantes do BMU são atualizados menos do que aqueles mais próximos do BMU. Isso preserva a estrutura topológica dos dados.
- Taxa de aprendizado: Uma taxa de aprendizado mais alta leva a atualizações maiores.
Esses passos são repetidos ao longo de muitas iterações, passando por muitos pontos de dados de entrada. Os pesos são gradualmente atualizados para que o mapa se auto-organize e capture a estrutura dos dados.
A taxa de aprendizado e o raio de vizinhança normalmente diminuem com o tempo, permitindo que o SOM ajuste os pesos gradualmente. Mais adiante no tutorial, implementamos os passos do processo de treinamento iterativo usando código Python.
Casos de uso típicos de SOMs
Dada sua arquitetura única, os SOMs têm várias aplicações típicas em aprendizado de máquina, análise e visualização.
Agrupamento
SOMs são usados para agrupar dados de alta dimensão em clusters de pontos de dados semelhantes. Isso ajuda a identificar estruturas inerentes dentro dos dados. Cada neurônio mapeia para pontos de dados intimamente relacionados e pontos de dados semelhantes são mapeados para os mesmos neurônios ou neurônios vizinhos, formando assim clusters distintos.
Por exemplo, no marketing, SOMs podem agrupar clientes com base no comportamento de compra. Isso permitiria que uma marca adaptasse estratégias de marketing para diferentes segmentos de clientes (clusters).
Redução de dimensionalidade
SOMs podem mapear dados de alta dimensão com muitos vetores de características para uma grade com menos dimensões (tipicamente, uma grade 2-dimensional). Como esse mapeamento preserva as relações entre os pontos de dados, a dimensionalidade reduzida facilita a visualização de conjuntos de dados complexos sem perda significativa de informações.
Além de facilitar as tarefas de visualização e análise, os SOMs também podem reduzir a dimensionalidade dos dados antes de aplicar outros algoritmos de aprendizado de máquina. Os SOMs também são utilizados no processamento de imagens para reduzir o número de características agrupando pixels ou regiões com características semelhantes, tornando as tarefas de reconhecimento e classificação de imagens mais eficientes.
Detecção de anomalias
Durante o processo de treinamento, a maioria dos pontos de dados é mapeada para neurônios específicos. Anomalias (pontos de dados que não se encaixam bem em nenhum cluster) são tipicamente mapeadas para neurônios distantes ou menos povoados. Esses outliers não se encaixam bem nos clusters aprendidos e são mapeados para neurônios distantes do BMU de pontos de dados normais.
Por exemplo, mapear transações financeiras em uma grade SOM nos permite identificar atividades incomuns (outliers) que podem indicar atividade potencialmente fraudulenta.
Visualização de dados
A visualização de dados é um dos casos de uso mais comuns dos SOMs. Dados de alta dimensão são difíceis de visualizar. Os SOMs facilitam a visualização ao reduzir a dimensionalidade dos dados e projetá-los em uma grade bidimensional. Isso também pode ajudar a detectar clusters nos dados e pontos de dados anômalos que seriam complicados de descobrir nos dados originais.
Por exemplo, a visualização de dados facilita a análise de dados populacionais de alta dimensão. Mapear os dados em uma grade 2D ajudaria a identificar semelhanças e diferenças entre diferentes grupos populacionais.
Depois de discutir os conceitos centrais e casos de uso dos SOMs, mostraremos como implementá-los usando o pacote Python MiniSom nas seções seguintes.
Você pode acessar e executar o código completo neste notebook do DataLab.
Configurando o Ambiente para SOM
Antes de construir o SOM, precisamos preparar o ambiente com os pacotes necessários.
Instalando bibliotecas Python
Precisamos destes pacotes:
- MiniSom é uma ferramenta Python baseada em NumPy que cria e treina SOMs.
- NumPy é utilizado para acessar funções matemáticas, como dividir arrays, obter valores únicos, etc.
matplotlibé utilizado para traçar vários gráficos e diagramas para visualizar os dados.- O pacote
datasetsdosklearné usado para importar conjuntos de dados nos quais aplicar o SOM. - O pacote
MinMaxScalerdosklearnnormaliza o conjunto de dados.
O seguinte trecho de código importa esses pacotes:
from minisom import MiniSom import numpy as np import matplotlib.pyplot as plt from sklearn import datasets from sklearn.preprocessing import MinMaxScaler
Preparando o conjunto de dados
Neste tutorial, usamos o MiniSom para construir um SOM e depois treiná-lo com o conjunto de dados IRIS. Este dataset consiste em 3 classes de plantas íris. Cada classe tem 50 instâncias. Para preparar os dados, seguimos estes passos:
- Importe o conjunto de dados Iris do
sklearn, - Extraia os vetores de dados e os escalares alvo.
- Normalize os vetores de dados. Neste tutorial, usamos o MinMaxScaler do scikit-learn.
- Declare um conjunto de rótulos para cada uma das três classes de plantas Iris.
O código a seguir implementa esses passos:
dataset_iris = datasets.load_iris() data_iris = dataset_iris.data target_iris = dataset_iris.target data_iris_normalized = MinMaxScaler().fit_transform(data_iris) labels_iris = {1:'1', 2:'2', 3:'3'} data = data_iris_normalized target = target_iris
Implementando Mapas Auto-Organizáveis (SOM) em Python
Para implementar um SOM em Python, definimos e inicializamos a grade antes de treiná-la no conjunto de dados. Podemos então visualizar os neurônios treinados e o conjunto de dados agrupado.
Definindo a grade SOM
Conforme explicado anteriormente, um SOM é uma grade de neurônios. Usando o MiniSom, podemos criar grades bidimensionais. As dimensões X e Y da grade são o número de neurônios ao longo de cada eixo. Para definir a grade SOM, também precisamos especificar:
- As dimensões X e Y da grade
- O número de variáveis de entrada – este é o número de linhas de dados.
Declare esses parâmetros como constantes Python:
SOM_X_AXIS_NODES = 8 SOM_Y_AXIS_NODES = 8 SOM_N_VARIABLES = data.shape[1]
O código de exemplo abaixo ilustra como declarar a grade usando o MiniSom:
som = MiniSom(SOM_X_AXIS_NODES, SOM_Y_AXIS_NODES, SOM_N_VARIABLES)
Os primeiros dois parâmetros são o número de neurônios ao longo dos eixos X e Y, e o terceiro parâmetro é o número de variáveis.
Declaramos outros parâmetros e hiperparâmetros ao criar a grade SOM. Explicaremos isso mais tarde no tutorial. Por enquanto, declare esses parâmetros conforme mostrado abaixo:
ALPHA = 0.5 DECAY_FUNC = 'linear_decay_to_zero' SIGMA0 = 1.5 SIGMA_DECAY_FUNC = 'linear_decay_to_one' NEIGHBORHOOD_FUNC = 'triangle' DISTANCE_FUNC = 'euclidean' TOPOLOGY = 'rectangular' RANDOM_SEED = 123
Crie um SOM usando esses parâmetros:
som = MiniSom( SOM_X_AXIS_NODES, SOM_Y_AXIS_NODES, SOM_N_VARIABLES, sigma=SIGMA0, learning_rate=ALPHA, neighborhood_function=NEIGHBORHOOD_FUNC, activation_distance=DISTANCE_FUNC, topology=TOPOLOGY, sigma_decay_function = SIGMA_DECAY_FUNC, decay_function = DECAY_FUNC, random_seed=RANDOM_SEED, )
Inicializando os neurônios
O comando acima cria um SOM com pesos aleatórios para todos os neurônios. Inicializar os neurônios com pesos extraídos dos dados (em vez de números aleatórios) pode tornar o processo de treinamento mais eficiente.
Ao usar o MiniSom para criar um Mapa Auto-Organizável (SOM), existem duas maneiras de inicializar os pesos dos neurônios com base nos dados:
- Inicialização aleatória: Os pesos iniciais dos neurônios são extraídos aleatoriamente dos dados de entrada. Fazemos isso aplicando a função
.random_weights_init()ao SOM. - Inicialização PCA: A inicialização pela Análise de Componentes Principais (PCA) utiliza os componentes principais dos dados de entrada para inicializar os pesos. Os pesos iniciais dos neurônios abrangem os dois primeiros componentes principais. Isso frequentemente leva a uma convergência mais rápida.
Neste guia, usamos a inicialização PCA. Para aplicar a inicialização PCA nos pesos do SOM, use a função .pca_weights_init() conforme mostrado abaixo:
som.pca_weights_init(data)
Treinando o SOM
O processo de treinamento atualiza os pesos do SOM para minimizar a distância entre os neurônios e os pontos de dados.
Abaixo, explicamos o processo iterativo de treinamento:
- Inicialização: Os vetores de peso de todos os neurônios são inicializados, tipicamente com valores aleatórios. Também é possível inicializar os pesos amostrando a distribuição dos dados de entrada.
- Seleção de entrada: Um vetor de entrada é (aleatoriamente) selecionado do conjunto de dados de treinamento.
- Identificação do BMU: O neurônio com o vetor de peso mais próximo do vetor de entrada é identificado como o BMU.
- Atualização do bairro: O BMU e seus neurônios vizinhos atualizam seus vetores de peso. A taxa de aprendizado e a função de vizinhança decidem quais neurônios são atualizados e em que medida. No passo de iteração t, dado o vetor de entrada x, o vetor de peso do neurônio i é wi, a taxa de aprendizado (t), e a função de vizinhança hbi (essa função quantifica a extensão da atualização para o neurônio i dada o neurônio BMU b), a fórmula de atualização de peso para o neurônio i é expressa como:

- Taxa de decaimento da taxa de aprendizado e do raio de vizinhança: Tanto a taxa de aprendizado quanto o raio de vizinhança diminuem ao longo do tempo. Nas iterações iniciais, o processo de treinamento faz ajustes maiores em uma vizinhança maior. Iterações posteriores ajudam a ajustar os pesos, fazendo mudanças menores nos pesos dos neurônios adjacentes. Isso permite que o mapa se estabilize e converja.
Para treinar o SOM, apresentamos ao modelo os dados de entrada. Podemos escolher uma das duas abordagens para fazer isso:
- Selecionar amostras aleatórias dos dados de entrada. A função
.train_random()implementa essa técnica. - Execute sequencialmente os vetores nos dados de entrada. Isso é feito usando a
.train_batch()função.
Essas funções aceitam os dados de entrada e o número de iterações como parâmetros. Neste guia, usamos a .train_random() função. Declare o número de iterações como uma constante e passe para a função de treinamento:
N_ITERATIONS = 5000 som.train_random(data, N_ITERATIONS, verbose=True)
Após executar o script e completar o treinamento, uma mensagem com o erro de quantização é exibida:
quantization error: 0.05357240680504421
O erro de quantização indica a quantidade de informação perdida quando o SOM quantiza (reduz a dimensionalidade) os dados. Um grande erro de quantização indica uma maior distância entre os neurônios e os pontos de dados. Isso também significa que a clusterização é menos confiável.
Visualizando neurônios do SOM
Agora temos um modelo SOM treinado. Para visualizá-lo, usamos um mapa de distâncias (também conhecido como um U-matriz). O mapa de distâncias exibe os neurônios do SOM como uma grade de células. A cor de cada célula representa sua distância em relação aos neurônios vizinhos.
O mapa de distâncias é uma grade com as mesmas dimensões do SOM. Cada célula no mapa de distâncias é a soma normalizada das distâncias (euclidianas) entre um neurônio e seus vizinhos.
Acesse o mapa de distâncias SOM usando a função .distance_map(). Para gerar a U-matriz, seguimos estes passos:
- Use
pyplotpara criar uma figura com as mesmas dimensões do SOM. Neste exemplo, as dimensões são 8×8. - Plote o mapa de distâncias usando matplotlib com a função
.pcolor(). Neste exemplo, usamosgist_yargcomo o esquema de cores. - Exiba a
colorbar, um índice que mapeia diferentes cores a diferentes valores escalares. Neste caso, como as distâncias estão normalizadas, os valores de distância escalar variam de 0 a 1.
O código abaixo implementa esses passos:
# crie a grade plt.figure(figsize=(8, 8)) # plote o mapa de distâncias plt.pcolor(som.distance_map().T, cmap='gist_yarg') # mostre a barra de cores plt.colorbar() plt.show()
Neste exemplo, a matriz U utiliza um esquema de cores monotônicas. Pode ser compreendida usando estas diretrizes:
- Tonais mais claros representam neurônios próximos, e tonais mais escuros representam neurônios mais distantes entre si.
- Grupos de tons mais claros podem ser interpretados como clusters. Nós escuros entre os clusters podem ser interpretados como as fronteiras entre os clusters.
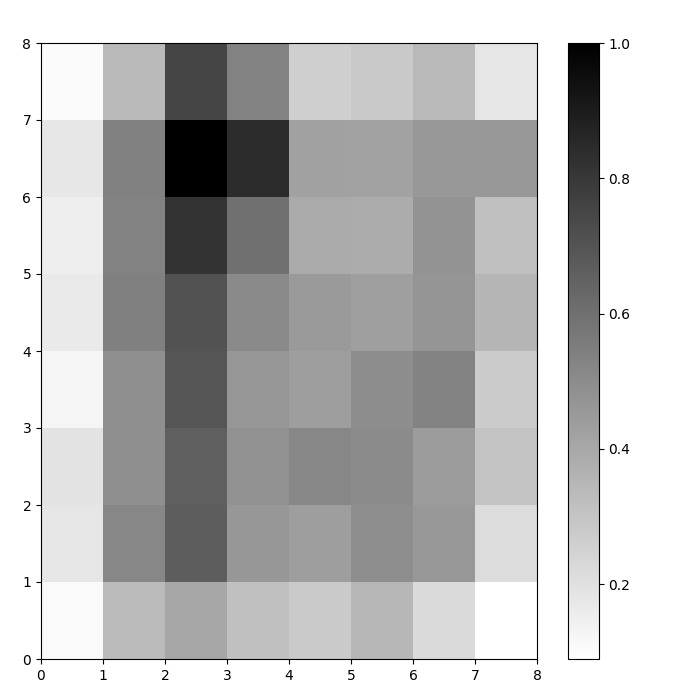
Figura 1: U-matrix de SOM treinado no conjunto de dados Iris (imagem do autor)
Avaliando os Resultados do Clustering SOM
A figura anterior ilustrou graficamente os neurônios do SOM. Nesta seção, mostramos como visualizar como o SOM agrupou os dados.
Identificando clusters
Sobrepomos marcadores sobre a U-matrix acima para denotar qual classe de planta Iris cada célula (neurônio) representa. Para fazer isso:
- Como antes, crie uma figura 8×8 usando
pyplot, plote o mapa de distâncias e mostre a barra de cores. - Especifique uma matriz de três marcadores matplotlib, um para cada classe de planta Iris.
- Especifique uma matriz de três códigos de cores matplotlib, um para cada classe de planta Iris.
- Plote iterativamente o neurônio vencedor para cada ponto de dados:
- Determine as (coordenadas do) neurônio vencedor para cada ponto de dados usando a função
.winner(). - Plote a posição de cada neurônio vencedor no meio de cada célula na grade.
w[0]ew[1]fornecem as coordenadas X e Y do neurônio, respectivamente. Um valor de 0,5 é adicionado a cada coordenada para plotá-la no meio da célula.
O código abaixo mostra como fazer isso:
# plote o mapa de distâncias plt.figure(figsize=(8, 8)) plt.pcolor(som.distance_map().T, cmap='gist_yarg') plt.colorbar() # crie os marcadores e cores para cada classe markers = ['o', 'x', '^'] colors = ['C0', 'C1', 'C2'] # plote o neurônio vencedor para cada ponto de dados for count, datapoint in enumerate(data): # obtenha o vencedor w = som.winner(datapoint) # coloque um marcador na posição vencedora para o ponto de dados amostral plt.plot(w[0]+.5, w[1]+.5, markers[target[count]-1], markerfacecolor='None', markeredgecolor=colors[target[count]-1], markersize=12, markeredgewidth=2) plt.show()
A imagem resultante é mostrada abaixo:
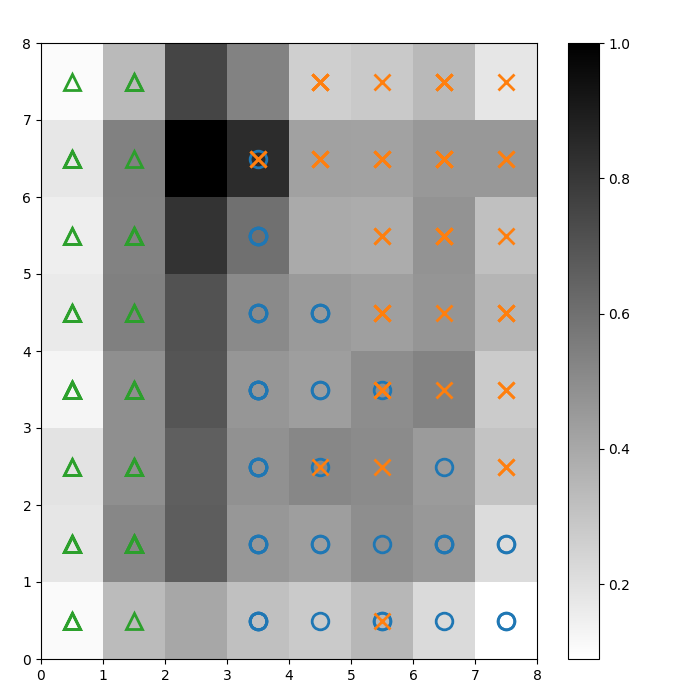
Figura 2: U-matrix sobreposta com marcadores de classe (imagem do autor)
Com base na documentação do conjunto de dados Iris, “uma classe é linearmente separável das outras 2; estas últimas não são linearmente separáveis entre si”. Na U-matrix acima, essas três classes são representadas por três marcadores – triângulo, círculo e cruz.
Observe que não há um limite claro entre os círculos azuis e as cruzes laranjas. Além disso, duas classes estão sobrepostas no mesmo neurônio em muitas células. Isso significa que o neurônio está equidistante de ambas as classes.
Visualizando o resultado da clusterização
Um SOM é um modelo de agrupamento. Pontos de dados semelhantes mapeiam para o mesmo neurônio. Pontos de dados da mesma classe mapeiam para um grupo de neurônios vizinhos. Nós plotamos todos os pontos de dados na grade do SOM para estudar melhor o comportamento de agrupamento.
Os seguintes passos descrevem como criar este gráfico de dispersão:
- Obtenha as coordenadas X e Y do neurônio vencedor para cada ponto de dados.
- Plote o mapa de distâncias, como fizemos para Figura 1.
- Use
plt.scatter()para fazer um gráfico de dispersão de todos os neurônios vencedores para cada ponto de dados. Adicione um deslocamento aleatório a cada ponto para evitar sobreposições entre os pontos de dados dentro da mesma célula.
Implementamos essas etapas no código abaixo:
# obtenha as coordenadas X e Y do neurônio vencedor para cada ponto de dadosw_x, w_y = zip(*[som.winner(d) for d in data]) w_x = np.array(w_x) w_y = np.array(w_y) # plote o mapa de distâncias plt.figure(figsize=(8, 8)) plt.pcolor(som.distance_map().T, cmap='gist_yarg', alpha=.2) plt.colorbar() # faça um gráfico de dispersão de todos os neurônios vencedores para cada ponto de dados # adicione um deslocamento aleatório a cada ponto para evitar sobreposições for c in np.unique(target): idx_target = target==c plt.scatter(w_x[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8, w_y[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8, s=50, c=colors[c-1], label=labels_iris[c+1] ) plt.legend(loc='upper right') plt.grid() plt.show()
O gráfico a seguir mostra o gráfico de dispersão resultante:
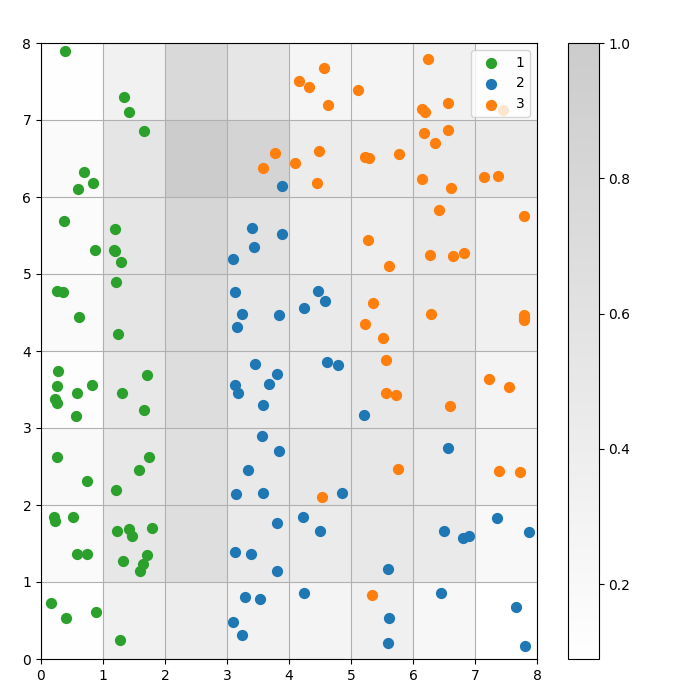 Figura 3: Gráfico de dispersão dos pontos de dados dentro das células (imagem do autor)
Figura 3: Gráfico de dispersão dos pontos de dados dentro das células (imagem do autor)
No gráfico de dispersão acima, observe que:
- Algumas células contêm tanto pontos azuis quanto laranja.
- Os pontos verdes estão claramente separados do resto dos dados, mas os pontos azuis e laranja não estão bem separados.
- As observações acima estão alinhadas com o fato de que apenas um dos três clusters no conjunto de dados Iris possui uma borda clara.
- No Figura 1, os nós escuros entre os clusters (que podem ser interpretados como as bordas entre os clusters) correspondem a células vazias no gráfico de dispersão.
Você pode acessar e executar o código completo neste notebook do DataLab.
Ajustando o Modelo SOM
As seções anteriores mostraram como criar e treinar um modelo SOM e como estudar os resultados visualmente. Nesta seção, discutimos como ajustar o desempenho dos modelos SOM.
Principais hiperparâmetros a serem ajustados
Assim como em qualquer modelo de aprendizado de máquina, os hiperparâmetros impactam consideravelmente o desempenho do modelo.
Alguns dos hiperparâmetros importantes no treinamento de SOMs são:
- O tamanho da grade determina o tamanho do mapa. O número de neurônios em um mapa com um tamanho de grade de AxB é A*B.
- A taxa de aprendizado decide quanto os pesos são alterados em cada iteração. Definimos a taxa de aprendizado inicial, e ela diminui ao longo do tempo de acordo com a função de decaimento.
- A função de decaimento decide a medida em que a taxa de aprendizado é diminuída em cada iteração subsequente.
- A função de vizinhança é uma função matemática que especifica quais neurônios devem ser considerados como vizinhos do BMU.
- A desvio padrão especifica a dispersão da função de vizinhança. Por exemplo, uma função de vizinhança gaussiana com um alto desvio padrão terá uma vizinhança maior do que a mesma função com um desvio padrão menor. Definimos o desvio padrão inicial, que diminui ao longo do tempo de acordo com a função de decaimento sigma.
- A decadência sigma controla o quanto o desvio padrão é reduzido em cada iteração subsequente.
- O número de iterações de treinamento decide quantas vezes os pesos são atualizados. Em cada iteração de treinamento, os pesos dos neurônios são atualizados uma vez.
- A função de distância é uma função matemática que calcula a distância entre neurônios e pontos de dados.
- A topologia decide o layout da estrutura da grade. Os neurônios na grade podem ser organizados em um padrão retangular ou hexagonal.
Na próxima seção, discutiremos diretrizes para definir os valores desses hiperparâmetros.
Impacto do ajuste de hiperparâmetros
Os valores dos hiperparâmetros devem ser decididos com base no modelo e no conjunto de dados. Até certo ponto, determinar esses valores é um processo de tentativa e erro. Nesta seção, fornecemos diretrizes para ajustar cada hiperparâmetro. Ao lado de cada hiperparâmetro, mencionamos (entre parênteses) as constantes Python respectivas usadas no código de exemplo.
- Tamanho da grade (
SOM_X_AXIS_NODESeSOM_X_AXIS_NODES): O tamanho da grade depende do tamanho do conjunto de dados. A regra geral é que, dado um conjunto de dados de tamanho N, a grade deve conter aproximadamente 5*sqrt(N) neurônios. Por exemplo, se o conjunto de dados tiver 150 amostras, a grade deve conter 5*sqrt(150) = aproximadamente 61 neurônios. Neste tutorial, o conjunto de dados Iris tem 150 linhas e usamos uma grade 8×8. - Taxa de aprendizado inicial (
ALPHA): Uma taxa mais alta acelera a convergência, enquanto taxas mais baixas são utilizadas para ajustes mais finos após as iterações iniciais. A taxa de aprendizado inicial deve ser grande o suficiente para permitir uma adaptação rápida, mas não tão grande a ponto de ultrapassar os valores ideais de peso. Neste artigo, a taxa de aprendizado inicial é 0,5. - Desvio padrão inicial (
SIGMA0): Ele determina o tamanho ou a dispersão inicial da vizinhança. Um valor maior considera mais padrões globais. Neste exemplo, usamos um desvio padrão inicial de 1,5. - Para a taxa de decaimento (
DECAY_FUNC) e a taxa de decaimento sigma (SIGMA_DECAY_FUNC), podemos escolher entre um dos três tipos de funções de decaimento: - Decaimento inverso: Esta função é adequada se os dados apresentam padrões globais e locais. Nesses casos, precisamos de uma fase mais longa de aprendizado amplo antes de nos concentrarmos nos padrões locais.
- Decaimento linear: Isso é bom para conjuntos de dados onde queremos um tamanho de vizinhança ou redução da taxa de aprendizado constante e uniforme. Isso é útil se os dados não precisarem de muito ajuste fino.
- Decaimento assintótico: Esta função é útil se os dados forem complexos e de alta dimensão. Nesses casos, é melhor gastar mais tempo na exploração global antes de fazer a transição gradual para detalhes mais finos.
- Função de vizinhança (
NEIGHBORHOOD_FUNC): A escolha padrão da função de vizinhança é a função Gaussiana. Outras funções, conforme explicado abaixo, também são utilizadas. - Gaussiana (padrão): Esta é uma curva em forma de sino. A medida em que um neurônio é atualizado diminui suavemente à medida que sua distância do neurônio vencedor aumenta. Ela proporciona uma transição suave e contínua e preserva a topologia dos dados. É adequada para a maioria dos propósitos gerais devido ao seu comportamento estável e previsível.
- Bolha: Esta função cria um vizinhança de largura fixa. Todos os neurônios dentro dessa vizinhança são atualizados igualmente, e neurônios fora dessa vizinhança não são atualizados (para um determinado ponto de dados). É computacionalmente mais barato e mais fácil de implementar. É útil para mapas menores onde limites de vizinhança nítidos não comprometem a agrupamento efetivo.
- Chapéu Mexicano: Ele tem uma região central positiva cercada por uma região negativa. Neurônios próximos ao BMU são atualizados para se aproximar do ponto de dados, e neurônios mais distantes são atualizados para se afastar do ponto de dados. Esta técnica realça o contraste e aguça as características do mapa. Como enfatiza clusters distintos, é eficaz em tarefas de reconhecimento de padrões onde uma separação clara dos clusters é desejada.
- Triângulo: Esta função define o tamanho do vizinhança como um triângulo, com o BMU tendo a maior influência. Ele diminui linearmente com a distância do BMU. É usado para agrupar dados com transições graduais entre grupos ou características, como dados de imagem, fala ou séries temporais, onde se espera que os pontos de dados vizinhos compartilhem características semelhantes.
- Função de distância (
DISTANCE_FUNC): Para medir a distância entre neurônios e pontos de dados, podemos escolher entre 4 métodos: - Distância Euclidiana (escolha padrão): Útil quando os dados são contínuos e queremos medir a distância em linha reta. É adequado para a maioria das tarefas gerais, especialmente quando os pontos de dados estão distribuídos uniformemente e relacionados espacialmente.
- Distância Coseno: Boa escolha para texto ou dados esparsos de alta dimensão, onde o ângulo entre os vetores é mais importante do que a magnitude. É útil para comparar a direcionalidade nos dados.
- Distância Manhattan: Ideal quando os pontos de dados estão em uma grade ou malha (por exemplo, quarteirões da cidade). Isso é menos sensível a outliers do que a distância Euclidiana.
- Distância de Chebyshev: Adequada para situações onde o movimento pode ocorrer em qualquer direção (por exemplo, distâncias em tabuleiros de xadrez). É útil para espaços discretos onde queremos priorizar a diferença máxima entre os eixos.
- Topologia (
TOPOLOGIA): Em uma grade, os neurônios podem ser organizados em uma estrutura hexagonal ou retangular: - Retangular (padrão): cada neurônio tem 4 vizinhos imediatos. Esta é a escolha certa quando os dados não têm uma relação espacial clara. É também computacionalmente mais simples.
- Hexagonal: cada neurônio tem 6 vizinhos. Esta é a opção preferida se os dados têm relações espaciais melhor representadas com uma grade hexagonal. Este é o caso de distribuições de dados circulares ou angulares.
- Número de iterações de treinamento (
N_ITERATIONS): Em princípio, tempos de treinamento mais longos levam a erros menores e melhor alinhamento dos pesos com os dados de entrada. No entanto, o desempenho do modelo aumenta assintoticamente com o número de iterações. Assim, após um certo número de iterações, o aumento de desempenho das interações subsequentes é apenas marginal. Decidir o número correto de iterações requer algum experimento. Neste tutorial, treinamos o modelo por 5000 iterações.
Para determinar a configuração correta de hiperparâmetros, recomendamos experimentar várias opções em um subconjunto menor dos dados.
Conclusão
Mapas auto-organizáveis são uma ferramenta robusta para aprendizado não supervisionado. Eles são usados para agrupamento, redução de dimensionalidade, detecção de anomalias e visualização de dados. Como preservam as propriedades topológicas de dados de alta dimensão e os representam em uma grade de baixa dimensão, os SOMs facilitam a visualização e interpretação de conjuntos de dados complexos.
Este tutorial discutiu os princípios subjacentes dos SOMs e mostrou como implementar um SOM usando a biblioteca MiniSom do Python. Também demonstrou como analisar visualmente os resultados e explicou os importantes hiperparâmetros utilizados para treinar os SOMs e ajustar seu desempenho.
Source:
https://www.datacamp.com/tutorial/self-organizing-maps













