Los mapas autoorganizados (SOM) son un tipo de red neuronal artificial utilizada para tareas de aprendizaje no supervisado como clustering. Dado un conjunto de puntos de datos, el modelo aprende a dividir los datos en clústeres. Los SOM proyectan datos complejos multidimensionales en una cuadrícula de menor dimensión (típicamente, 2 dimensiones). Esto facilita una visualización más sencilla. Por lo tanto, los SOM también se utilizan para visualizar datos de alta dimensión.
En este tutorial, exploramos los conceptos básicos de los SOM, su proceso de aprendizaje y sus casos de uso típicos. Explicamos cómo implementar un SOM en Python utilizando la biblioteca MiniSom y cómo representar los resultados visualmente. Finalmente, discutimos parámetros hiperimportantes en el entrenamiento de modelos SOM y cómo ajustarlos.
Entendiendo los Mapas Autoorganizados (SOM)
En esta sección, presentamos los conceptos básicos de los MAPs, su proceso de aprendizaje y sus casos de uso.
Conceptos básicos de los MAPs
Los mapas autoorganizados implican un conjunto de conceptos que son importantes de entender.
Neurones
Un MAP es esencialmente una cuadrícula de neuronas. Durante el entrenamiento, la neurona cuyo vector de pesos está más cercano a un punto de datos de entrada ajusta sus pesos y los pesos de sus vecinos para coincidir aún más con el punto de datos de entrada. A lo largo de muchas iteraciones, grupos de neuronas vecinas se asignan a puntos de datos relacionados. Esto resulta en la agrupación del conjunto de datos de entrada.
Estructura de cuadrícula
Las neuronas en un SOM están organizadas como una cuadrícula. Las neuronas vecinas se asignan a puntos de datos similares. Los conjuntos de datos más grandes necesitan una cuadrícula más grande. Típicamente, esta cuadrícula es bidimensional. La estructura de la cuadrícula sirve como el espacio de baja dimensión al que se asignan datos de alta dimensión. La cuadrícula ayuda a visualizar patrones y grupos de datos.
Asignación de puntos de datos
Cada punto de datos se compara con todas las neuronas utilizando una métrica de distancia. La neurona cuyo vector de peso esté más cercano al punto de datos de entrada es la Unidad de Mejor Coincidencia (BMU) para ese punto de datos.
Una vez que se identifica el BMU, se actualizan los pesos del BMU y de sus neuronas vecinas. Esta actualización acerca aún más al BMU y a las neuronas vecinas al punto de datos de entrada. Este mapeo preserva la topología de los datos, asegurando que puntos de datos similares se mapeen a neuronas cercanas.
Proceso de aprendizaje de SOMs
El entrenamiento de SOM se considera no supervisado porque no se basa en conjuntos de datos etiquetados. El objetivo del entrenamiento de SOM es ajustar iterativamente los vectores de peso de las neuronas para que puntos de datos similares se mapeen a neuronas cercanas.
Aprendizaje competitivo
Las SOMs utilizan el aprendizaje competitivo (en lugar del descenso de gradiente y la retropropagación). Las neuronas compiten para convertirse en la Unidad de Mejor Coincidencia (BMU) para cada punto de datos de entrada. La neurona más cercana a un punto de datos se determina como su BMU. La BMU y sus neuronas vecinas se actualizan para reducir aún más su distancia al punto de datos. Las neuronas vecinas se mapean a puntos de datos relacionados. Esto conduce a la especialización entre las neuronas y la agrupación de los puntos de datos de entrada.
Funciones de distancia
Las SOMs utilizan una función de distancia para medir la distancia entre neuronas y puntos de datos. Esta distancia se utiliza para determinar la BMU de cada punto de datos. MiniSom tiene cuatro funciones de distancia para elegir:
- distancia euclidiana (esta es la opción predeterminada)
- Distancia coseno
- Distancia Manhattan
- Distancia Chebyshev
Función de vecindario
Después de identificar el BMU de un punto de datos, el BMU y sus neuronas vecinas se actualizan en la dirección de ese punto de datos. La función de vecindario asegura que el SOM mantenga las relaciones topológicas de los datos de entrada. La función de vecindario decide:
- Qué neuronas se consideran en el vecindario del BMU
- El grado en que se actualizan las neuronas vecinas. En general, las neuronas más cercanas al BMU reciben un ajuste mayor que aquellas más alejadas
MiniSom viene con tres funciones de vecindario:
- Gaussiana (esta es la predeterminada).
- Bola
- Sombrero mexicano
- Triángulo
Proceso iterativo
El proceso de aprendizaje en los SOMs ocurre a lo largo de varias iteraciones. En cada iteración, el SOM procesa muchos puntos de datos de entrada. Esbozamos el proceso de aprendizaje en los siguientes pasos:
- Cuando comienza el entrenamiento, los pesos de todas las neuronas se inicializan (aleatoriamente).
- Dado un punto de datos de entrada, cada neurona calcula su distancia desde la entrada.
- La neurona con la distancia más pequeña se declara el BMU.
- Los pesos del BMU y sus vecinos se ajustan para acercarse al vector de entrada. La magnitud de este ajuste se decide por:
- Una función de vecindad: Las neuronas más alejadas del BMU se actualizan menos que aquellas más cercanas al BMU. Esto preserva la estructura topológica de los datos.
- Tasa de aprendizaje: Una tasa de aprendizaje más alta lleva a actualizaciones más grandes.
Estos pasos se repiten a lo largo de muchas iteraciones, pasando por muchos puntos de datos de entrada. Los pesos se actualizan gradualmente para que el mapa se autoorganice y capture la estructura de los datos.
La tasa de aprendizaje y el radio del vecindario típicamente disminuyen con el tiempo, permitiendo que el SOM ajuste gradualmente los pesos. Más adelante en el tutorial, implementamos los pasos del proceso de entrenamiento iterativo utilizando código Python.
Casos de uso típicos de los SOM
Dada su arquitectura única, los SOM tienen varias aplicaciones típicas en aprendizaje automático, análisis y visualización.
Agrupamiento
Los SOMs se utilizan para agrupar datos de alta dimensión en clústeres de puntos de datos similares. Esto ayuda a identificar estructuras inherentes dentro de los datos. Cada neurona mapea a puntos de datos estrechamente relacionados y los puntos de datos similares se mapean a la misma neurona o a neuronas vecinas, formando así clústeres distintos.
Por ejemplo, en marketing, los SOMs pueden agrupar a los clientes según su comportamiento de compra. Esto permitiría a una marca adaptar estrategias de marketing a diferentes segmentos de clientes (clústeres).
Reducción de dimensionalidad
Los SOMs pueden mapear datos de alta dimensión con muchos vectores de características a una cuadrícula con menos dimensiones (típicamente, una cuadrícula de 2 dimensiones). Dado que este mapeo preserva las relaciones entre los puntos de datos, la reducción de dimensionalidad facilita la visualización de conjuntos de datos complejos sin pérdida significativa de información.
Además de facilitar las tareas de visualización y análisis, los SOM también pueden reducir la dimensionalidad de los datos antes de aplicar otros algoritmos de aprendizaje automático. Los SOM también se utilizan en el procesamiento de imágenes para reducir el número de características agrupando píxeles o regiones con características similares, lo que hace que las tareas de reconocimiento y clasificación de imágenes sean más eficientes.
Detección de anomalías
Durante el proceso de entrenamiento, la mayoría de los puntos de datos se mapean a neuronas específicas. Las anomalías (puntos de datos que no encajan bien en ningún clúster) suelen mapearse a neuronas distantes o menos pobladas. Estos valores atípicos no encajan bien en los clústeres aprendidos y se mapean a neuronas alejadas de la BMU de los puntos de datos normales.
Por ejemplo, mapear transacciones financieras en una cuadrícula SOM nos permite identificar actividades inusuales (valores atípicos) que pueden indicar una actividad potencialmente fraudulenta.
Visualización de datos
La visualización de datos es uno de los casos de uso más comunes de los SOM. Los datos de alta dimensión son difíciles de visualizar. Los SOM facilitan la visualización al reducir la dimensionalidad de los datos y proyectarlos en una cuadrícula bidimensional. Esto también puede ayudar a detectar grupos en los datos y puntos de datos anómalos que serían complicados de descubrir en los datos originales.
Por ejemplo, la visualización de datos facilita el análisis de datos poblacionales de alta dimensión. Mapear los datos en una cuadrícula 2D ayudaría a identificar similitudes y diferencias entre diferentes grupos poblacionales.
Después de discutir los conceptos básicos y casos de uso de los SOM, mostraremos cómo implementarlos utilizando el paquete de Python MiniSom en las siguientes secciones.
Puedes acceder y ejecutar el código completo en este cuaderno de DataLab.
Configurando el Entorno para SOM
Antes de construir el SOM, necesitamos preparar el entorno con los paquetes necesarios.
Instalación de bibliotecas de Python
Necesitamos estos paquetes:
- MiniSom es una herramienta de Python basada en NumPy que crea y entrena SOMs.
- NumPy se utiliza para acceder a funciones matemáticas como dividir arreglos, obtener valores únicos, etc.
matplotlibse utiliza para trazar varios gráficos y diagramas para visualizar los datos.- El paquete
datasetsdesklearnse utiliza para importar conjuntos de datos sobre los que aplicar el SOM. - El paquete
MinMaxScalerdesklearnnormaliza el conjunto de datos.
El siguiente fragmento de código importa estos paquetes:
from minisom import MiniSom import numpy as np import matplotlib.pyplot as plt from sklearn import datasets from sklearn.preprocessing import MinMaxScaler
Preparando el conjunto de datos
En este tutorial, usamos MiniSom para construir un SOM y luego entrenarlo en el conjunto de datos IRIS. Este dataset consiste en 3 clases de plantas de iris. Cada clase tiene 50 instancias. Para preparar los datos, seguimos estos pasos:
- Importar el conjunto de datos Iris desde
sklearn, - Extraer los vectores de datos y los escalares objetivo.
- Normaliza los vectores de datos. En este tutorial, utilizamos el MinMaxScaler de scikit-learn.
- Declara un conjunto de etiquetas para cada una de las tres clases de plantas Iris.
El siguiente código implementa estos pasos:
dataset_iris = datasets.load_iris() data_iris = dataset_iris.data target_iris = dataset_iris.target data_iris_normalized = MinMaxScaler().fit_transform(data_iris) labels_iris = {1:'1', 2:'2', 3:'3'} data = data_iris_normalized target = target_iris
Implementación de Mapas Auto-Organizados (SOM) en Python
Para implementar un SOM en Python, definimos e inicializamos la cuadrícula antes de entrenarla en el conjunto de datos. Luego podemos visualizar las neuronas entrenadas y el conjunto de datos agrupado.
Definiendo la cuadrícula SOM
Como se explicó anteriormente, un SOM es una cuadrícula de neuronas. Usando MiniSom, podemos crear cuadrículas de 2 dimensiones. Las dimensiones X e Y de la cuadrícula son el número de neuronas a lo largo de cada eje. Para definir la cuadrícula SOM, también necesitamos especificar:
- Las dimensiones X e Y de la cuadrícula
- El número de variables de entrada – este es el número de filas de datos.
Declara estos parámetros como constantes de Python:
SOM_X_AXIS_NODES = 8 SOM_Y_AXIS_NODES = 8 SOM_N_VARIABLES = data.shape[1]
El código de muestra a continuación ilustra cómo declarar la cuadrícula usando MiniSom:
som = MiniSom(SOM_X_AXIS_NODES, SOM_Y_AXIS_NODES, SOM_N_VARIABLES)
Los primeros dos parámetros son el número de neuronas a lo largo de los ejes X e Y, y el tercer parámetro es el número de variables.
Declaramos otros parámetros e hiperpárametros al crear la cuadrícula SOM. Explicaremos estos más adelante en el tutorial. Por ahora, declara estos parámetros como se muestra a continuación:
ALPHA = 0.5 DECAY_FUNC = 'linear_decay_to_zero' SIGMA0 = 1.5 SIGMA_DECAY_FUNC = 'linear_decay_to_one' NEIGHBORHOOD_FUNC = 'triangle' DISTANCE_FUNC = 'euclidean' TOPOLOGY = 'rectangular' RANDOM_SEED = 123
Crea un SOM usando estos parámetros:
som = MiniSom( SOM_X_AXIS_NODES, SOM_Y_AXIS_NODES, SOM_N_VARIABLES, sigma=SIGMA0, learning_rate=ALPHA, neighborhood_function=NEIGHBORHOOD_FUNC, activation_distance=DISTANCE_FUNC, topology=TOPOLOGY, sigma_decay_function = SIGMA_DECAY_FUNC, decay_function = DECAY_FUNC, random_seed=RANDOM_SEED, )
Inicializando las neuronas
El comando anterior crea un SOM con pesos aleatorios para todas las neuronas. Inicializar las neuronas con pesos extraídos de los datos (en lugar de números aleatorios) puede hacer que el proceso de entrenamiento sea más eficiente.
Al usar MiniSom para crear un Mapa Auto-Organizado (SOM), hay dos formas de inicializar los pesos de las neuronas basándose en los datos:
- Inicialización aleatoria: Los pesos iniciales de las neuronas se extraen aleatoriamente de los datos de entrada. Hacemos esto aplicando la función
.random_weights_init()al SOM. - Inicialización PCA: La inicialización mediante Análisis de Componentes Principales (PCA) utiliza los componentes principales de los datos de entrada para inicializar los pesos. Los pesos iniciales de las neuronas abarcan los primeros dos componentes principales. Esto a menudo conduce a una convergencia más rápida.
En esta guía, utilizamos la inicialización PCA. Para aplicar la inicialización PCA en los pesos del SOM, utiliza la .pca_weights_init() función como se muestra a continuación:
som.pca_weights_init(data)
Entrenando el SOM
El proceso de entrenamiento actualiza los pesos del SOM para minimizar la distancia entre las neuronas y los puntos de datos.
A continuación, explicamos el proceso de entrenamiento iterativo:
- Inicialización: Los vectores de peso de todas las neuronas se inicializan, típicamente con valores aleatorios. También es posible inicializar los pesos muestreando la distribución de datos de entrada.
- Selección de entrada: Se selecciona (aleatoriamente) un vector de entrada del conjunto de datos de entrenamiento.
- Identificación de BMU: Se identifica la neurona cuyo vector de peso es el más cercano al vector de entrada como la BMU.
- Actualización del vecindario: La BMU y sus neuronas vecinas actualizan sus vectores de peso. La tasa de aprendizaje y la función de vecindario deciden qué neuronas se actualizan y en qué medida. En el paso de iteración t, dado el vector de entrada x, el vector de peso de la neurona i es wi, la tasa de aprendizaje (t), y la función de vecindario hbi (esta función cuantifica la extensión de la actualización para la neurona i dado la neurona BMU b), la fórmula de actualización de peso para la neurona i se expresa como:

- Tasa de decaimiento de la tasa de aprendizaje y el radio de vecindario: Tanto la tasa de aprendizaje como el radio de vecindario disminuyen con el tiempo. En las primeras iteraciones, el proceso de entrenamiento realiza ajustes más grandes sobre un vecindario más amplio. Las iteraciones posteriores ayudan a afinar los pesos realizando cambios más pequeños en los pesos de las neuronas adyacentes. Esto permite que el mapa se estabilice y converja.
Para entrenar el SOM, presentamos al modelo los datos de entrada. Podemos elegir entre uno de dos enfoques para hacerlo:
- Seleccionar muestras al azar de los datos de entrada. La
.train_random()función implementa esta técnica. - Ejecutar secuencialmente los vectores en los datos de entrada. Esto se hace utilizando la función
.train_batch()función.
Estas funciones aceptan los datos de entrada y el número de iteraciones como parámetros. En esta guía, utilizamos la función .train_random() función. Declara el número de iteraciones como una constante y pásalo a la función de entrenamiento:
N_ITERATIONS = 5000 som.train_random(data, N_ITERATIONS, verbose=True)
Después de ejecutar el script y completar el entrenamiento, se muestra un mensaje con el error de cuantización:
quantization error: 0.05357240680504421
El error de cuantización indica la cantidad de información perdida cuando el SOM cuantiza (reduce la dimensionalidad de) los datos. Un gran error de cuantización indica una mayor distancia entre las neuronas y los puntos de datos. También significa que el agrupamiento es menos confiable.
Visualizando neuronas SOM
Ahora tenemos un modelo SOM entrenado. Para visualizarlo, usamos un mapa de distancias (también conocido como un U-matriz). El mapa de distancias muestra las neuronas del SOM como una cuadrícula de celdas. El color de cada celda representa su distancia a las neuronas vecinas.
El mapa de distancias es una cuadrícula con las mismas dimensiones que el SOM. Cada celda en el mapa de distancias es la suma normalizada de las distancias (euclidianas) entre una neurona y sus vecinas.
Accede al mapa de distancias SOM utilizando la función .distance_map(). Para generar la U-matriz, seguimos estos pasos:
- Usa
pyplotpara crear una figura con las mismas dimensiones que el SOM. En este ejemplo, las dimensiones son 8×8. - Dibuja el mapa de distancias utilizando matplotlib con la función
.pcolor()En este ejemplo, usamosgist_yargcomo esquema de color. - Muestra la
barra de colores, un mapeo de índices que asigna diferentes colores a diferentes valores escalares. En este caso, dado que las distancias están normalizadas, los valores escalares de distancia van de 0 a 1.
El código a continuación implementa estos pasos:
# crear la cuadrícula plt.figure(figsize=(8, 8)) # trazar el mapa de distancia plt.pcolor(som.distance_map().T, cmap='gist_yarg') # mostrar la barra de colores plt.colorbar() plt.show()
En este ejemplo, la U-matriz utiliza un esquema de colores monotono. Se puede entender siguiendo estas pautas:
- Los tonos más claros representan neuronas cercanas entre sí, y los tonos más oscuros representan neuronas más alejadas de otras.
- Grupos de tonos más claros pueden interpretarse como clústeres. Los nodos oscuros entre los clústeres pueden interpretarse como los límites entre clústeres.
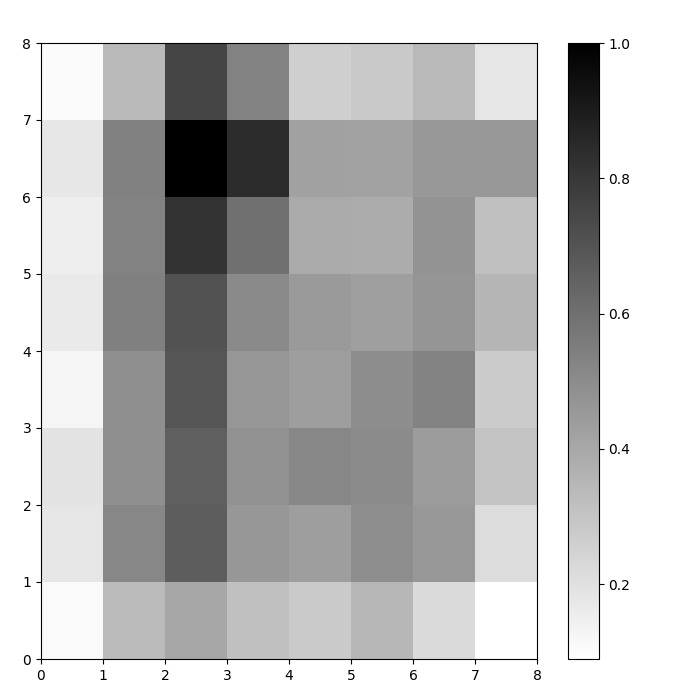
Figura 1: U-matriz de SOM entrenada con el conjunto de datos Iris (imagen del autor)
Evaluando los Resultados de Clustering de SOM
La figura anterior ilustró gráficamente las neuronas del SOM. En esta sección, mostramos cómo visualizar cómo el SOM agrupó los datos.
Identificando clústeres
Superponemos marcadores sobre la U-matriz anterior para denotar a qué clase de planta Iris representa cada celda (neurona). Para hacer esto:
- Como antes, crea una figura de 8×8 usando
pyplot, traza el mapa de distancias y muestra la barra de color. - Especifica un array de tres marcadores de matplotlib, uno para cada clase de planta Iris.
- Especifica un array de tres códigos de color de matplotlib, uno para cada clase de planta Iris.
- Traza de manera iterativa el neurona ganadora para cada punto de datos:
- Determina las (coordenadas del) neurona ganadora para cada punto de datos usando la
.winner() función. - Plotea la posición de cada neurona ganadora en el medio de cada celda en la cuadrícula.
w[0]yw[1]dan las coordenadas X e Y de la neurona, respectivamente. Se agrega un valor de 0.5 a cada coordenada para ploteada en el medio de la celda.
El código a continuación muestra cómo hacer esto:
# plotea el mapa de distancias plt.figure(figsize=(8, 8)) plt.pcolor(som.distance_map().T, cmap='gist_yarg') plt.colorbar() # crea los marcadores y colores para cada clase markers = ['o', 'x', '^'] colors = ['C0', 'C1', 'C2'] # plotea la neurona ganadora para cada punto de datos for count, datapoint in enumerate(data): # obtiene la ganadora w = som.winner(datapoint) # coloca un marcador en la posición ganadora para el punto de datos de muestra plt.plot(w[0]+.5, w[1]+.5, markers[target[count]-1], markerfacecolor='None', markeredgecolor=colors[target[count]-1], markersize=12, markeredgewidth=2) plt.show()
La imagen resultante se muestra a continuación:
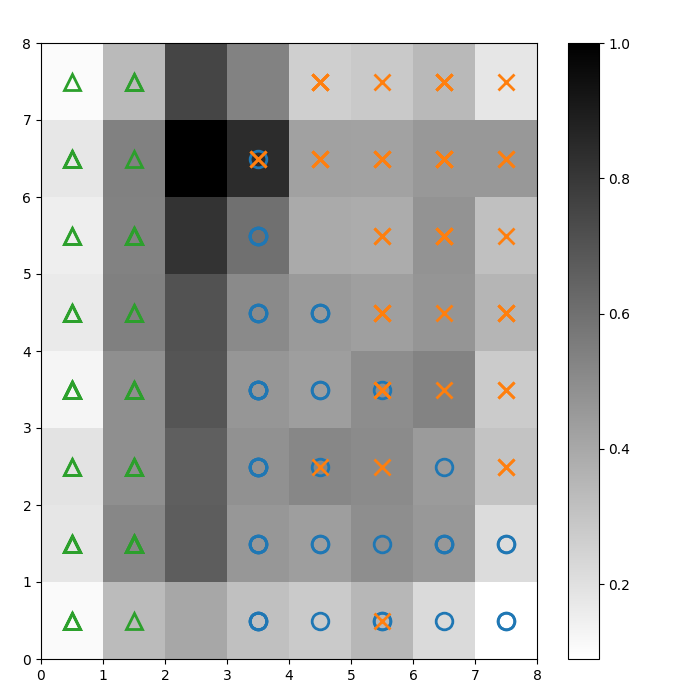
Figura 2: U-matriz superpuesta con marcadores de clase (imagen del autor)
Basado en la documentación del conjunto de datos Iris, “una clase es linealmente separable de las otras 2; las últimas no son linealmente separables entre sí”. En la U-matriz anterior, estas tres clases están representadas por tres marcadores: triángulo, círculo y cruz.
Nota que no hay un límite claro entre los círculos azules y las cruces naranjas. Además, dos clases están superpuestas en la misma neurona en muchas celdas. Esto significa que la neurona está a la misma distancia de ambas clases.
Visualizando el resultado del agrupamiento
Un SOM es un modelo de agrupamiento. Los puntos de datos similares se mapean al mismo neurona. Los puntos de datos de la misma clase se mapean a un clúster de neuronas vecinas. Graficamos todos los puntos de datos en la cuadrícula del SOM para estudiar mejor el comportamiento de agrupamiento.
Los siguientes pasos describen cómo crear este diagrama de dispersión:
- Obtén las coordenadas X e Y de la neurona ganadora para cada punto de datos.
- Grafica el mapa de distancias, como hicimos para la Figura 1.
- Utiliza
plt.scatter()para hacer un diagrama de dispersión de todas las neuronas ganadoras para cada punto de datos. Añade un desplazamiento aleatorio a cada punto para evitar superposiciones entre los puntos de datos dentro de la misma celda.
Implementamos estos pasos en el código a continuación:
# obtener las coordenadas X e Y de la neurona ganadora para cada punto de datosw_x, w_y = zip(*[som.winner(d) for d in data]) w_x = np.array(w_x) w_y = np.array(w_y) # trazar el mapa de distancias plt.figure(figsize=(8, 8)) plt.pcolor(som.distance_map().T, cmap='gist_yarg', alpha=.2) plt.colorbar() # hacer un diagrama de dispersión de todas las neuronas ganadoras para cada punto de datos # añadir un desplazamiento aleatorio a cada punto para evitar superposiciones for c in np.unique(target): idx_target = target==c plt.scatter(w_x[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8, w_y[idx_target]+.5+(np.random.rand(np.sum(idx_target))-.5)*.8, s=50, c=colors[c-1], label=labels_iris[c+1] ) plt.legend(loc='upper right') plt.grid() plt.show()
El siguiente gráfico muestra el diagrama de dispersión resultante:
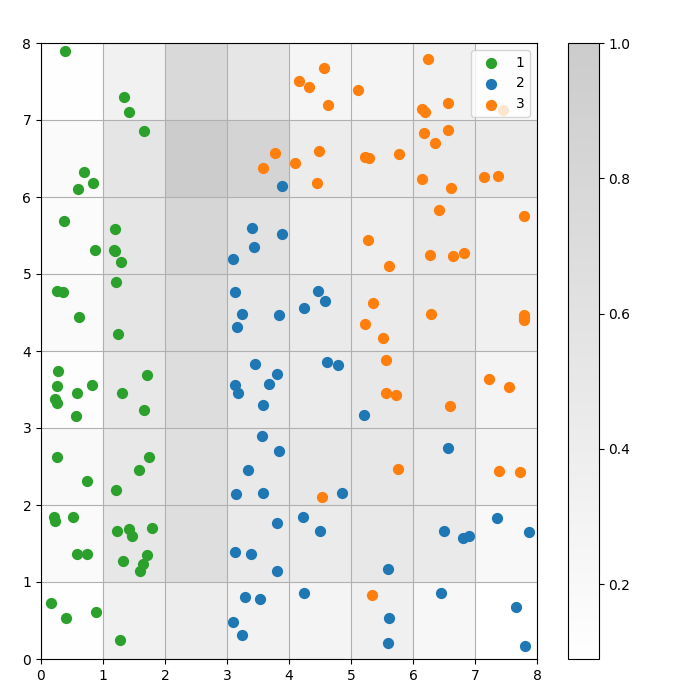 Figura 3: Gráfico de dispersión de puntos de datos dentro de las celdas (imagen del autor)
Figura 3: Gráfico de dispersión de puntos de datos dentro de las celdas (imagen del autor)
En el gráfico de dispersión anterior, observa que:
- Algunas celdas contienen tanto puntos azules como naranjas.
- Los puntos verdes están claramente separados del resto de los datos, pero los puntos azules y naranjas no están separados de manera limpia.
- Las observaciones anteriores coinciden con el hecho de que solo uno de los tres grupos en el conjunto de datos de Iris tiene un límite claro.
- En Figura 1, los nodos oscuros entre los grupos (que pueden interpretarse como los límites entre grupos) coinciden con celdas vacías en el gráfico de dispersión.
Puedes acceder y ejecutar el código completo en este cuaderno de DataLab.
Ajuste del Modelo SOM
Las secciones anteriores mostraron cómo crear y entrenar un modelo SOM y cómo estudiar los resultados visualmente. En esta sección, discutimos cómo ajustar el rendimiento de los modelos SOM.
Parámetros hiperimportantes a ajustar
Al igual que con cualquier modelo de aprendizaje automático, los hiperparámetros impactan considerablemente en el rendimiento del modelo.
Algunos de los hiperparámetros importantes en el entrenamiento de SOMs son:
- El tamaño de la cuadrícula decide el tamaño del mapa. El número de neuronas en un mapa con un tamaño de cuadrícula de AxB es A*B.
- La tasa de aprendizaje decide cuánto se cambian los pesos en cada iteración. Establecemos la tasa de aprendizaje inicial, y esta disminuye con el tiempo según la función de decaimiento.
- La función de decaimiento decide el grado en que se disminuye la tasa de aprendizaje en cada iteración subsiguiente.
- La función de vecindario es una función matemática que especifica qué neuronas se consideran como los vecinos del BMU.
- La desviación estándar especifica la extensión de la función de vecindario. Por ejemplo, una función de vecindario gaussiana con una alta desviación estándar tendrá un vecindario más grande que la misma función con una desviación estándar más pequeña. Establecemos la desviación estándar inicial, que disminuye con el tiempo de acuerdo con la función de decaimiento sigma.
- La función de decaimiento sigma controla cuánto se reduce la desviación estándar en cada iteración subsiguiente.
- El número de iteraciones de entrenamiento decide cuántas veces se actualizan los pesos. En cada iteración de entrenamiento, los pesos de las neuronas se actualizan una vez.
- La función de distancia es una función matemática que calcula la distancia entre neuronas y puntos de datos.
- La topología decide la disposición de la estructura de la cuadrícula. Las neuronas en la cuadrícula pueden disponerse en un patrón rectangular o hexagonal.
En la siguiente sección, discutimos pautas para establecer los valores de estos hiperparámetros.
Impacto de la sintonización de hiperparámetros
Los valores de los hiperparámetros deben decidirse en función del modelo y del conjunto de datos. Hasta cierto punto, determinar estos valores es un proceso de prueba y error. En esta sección, ofrecemos pautas para ajustar cada hiperparámetro. Junto a cada hiperparámetro, mencionamos (entre paréntesis) las constantes de Python respectivas utilizadas en el código de ejemplo.
- Tamaño de la cuadrícula (
SOM_X_AXIS_NODESySOM_X_AXIS_NODES): El tamaño de la cuadrícula depende del tamaño del conjunto de datos. La regla general es que dado un conjunto de datos de tamaño N, la cuadrícula debe contener aproximadamente 5*sqrt(N) neuronas. Por ejemplo, si el conjunto de datos tiene 150 muestras, la cuadrícula debe contener 5*sqrt(150) = aproximadamente 61 neuronas. En este tutorial, el conjunto de datos Iris tiene 150 filas y utilizamos una cuadrícula de 8×8. - Tasa de aprendizaje inicial (
ALPHA): Una tasa más alta acelera la convergencia, mientras que tasas más bajas se utilizan para ajustes más finos después de las iteraciones iniciales. La tasa de aprendizaje inicial debe ser lo suficientemente grande como para permitir una rápida adaptación, pero no tan grande que exceda los valores óptimos de peso. En este artículo, la tasa de aprendizaje inicial es 0.5. - Desviación estándar inicial (
SIGMA0): Determina el tamaño o la dispersión inicial del vecindario. Un valor más grande considera patrones más globales. En este ejemplo, usamos una desviación estándar inicial de 1.5. - Para la tasa de descomposición (
DECAY_FUNC) y la tasa de descomposición sigma (SIGMA_DECAY_FUNC), podemos elegir entre uno de tres tipos de funciones de descomposición: - Decaimiento inverso: Esta función es adecuada si los datos tienen patrones globales y locales. En tales casos, necesitamos una fase más larga de aprendizaje amplio antes de centrarnos en los patrones locales.
- Decaimiento lineal: Esto es bueno para conjuntos de datos donde queremos un tamaño de vecindario constante y uniforme o una reducción en la tasa de aprendizaje. Esto es útil si los datos no necesitan mucho ajuste fino.
- Decaimiento asintótico: Esta función es útil si los datos son complejos y de alta dimensión. En tales casos, es mejor dedicar más tiempo a la exploración global antes de pasar gradualmente a detalles más finos.
- Función de vecindad (
NEIGHBORHOOD_FUNC): La elección predeterminada de la función de vecindad es la función gaussiana. Otras funciones, como se explica a continuación, también se utilizan. - Gaussiana (predeterminada): Esta es una curva en forma de campana. La medida en que se actualiza una neurona disminuye suavemente a medida que aumenta su distancia desde la neurona ganadora. Proporciona una transición suave y continua y preserva la topología de los datos. Es adecuada para la mayoría de los propósitos generales debido a su comportamiento estable y predecible.
- Burbuja: Esta función crea un vecindario de ancho fijo. Todas las neuronas dentro de este vecindario se actualizan por igual, y las neuronas fuera de este vecindario no se actualizan (para un punto de datos dado). Es computacionalmente más barato y más fácil de implementar. Es útil para mapas más pequeños donde los límites del vecindario definidos no comprometen el agrupamiento efectivo.
- Sombrero mexicano: Tiene una región central positiva rodeada por una región negativa. Las neuronas cercanas a la BMU se actualizan para acercarse al punto de datos, y las neuronas más alejadas se actualizan para alejarse del punto de datos. Esta técnica mejora el contraste y agudiza las características en el mapa. Dado que enfatiza los clústeres distintos, es efectiva en tareas de reconocimiento de patrones donde se desea una clara separación de clústeres.
- Triángulo: Esta función define el tamaño del vecindario como un triángulo, donde el BMU tiene la mayor influencia. Disminuye linealmente con la distancia desde el BMU. Se utiliza para agrupar datos con transiciones graduales entre clústeres o características, como datos de imagen, habla o series temporales, donde se espera que los puntos de datos vecinos compartan características similares.
- Función de distancia (
DISTANCE_FUNC): Para medir la distancia entre neuronas y puntos de datos, podemos elegir entre 4 métodos: - Distancia euclidiana (opción predeterminada): Útil cuando los datos son continuos y queremos medir la distancia en línea recta. Se adapta a la mayoría de las tareas generales, especialmente cuando los puntos de datos están distribuidos uniformemente y son espacialmente relacionados.
- Distancia coseno: Buen opción para datos de texto o de alta dimensión y dispersos donde el ángulo entre los vectores es más importante que la magnitud. Es útil para comparar la direccionalidad en los datos.
- Distancia de Manhattan: Ideal cuando los puntos de datos están en una cuadrícula o red (por ejemplo, bloques de la ciudad). Esto es menos sensible a los valores atípicos que la distancia euclidiana.
- Distancia de Chebyshev: Adecuada para situaciones donde el movimiento puede ocurrir en cualquier dirección (por ejemplo, distancias en un tablero de ajedrez). Es útil para espacios discretos donde queremos priorizar la diferencia máxima en el eje.
- Topología (
TOPOLOGY): En una cuadrícula, las neuronas pueden estar dispuestas en una estructura hexagonal o rectangular: - Rectangular (predeterminado): cada neurona tiene 4 vecinos inmediatos. Esta es la opción correcta cuando los datos no tienen una relación espacial clara. También es computacionalmente más simple.
- Hexagonal: cada neurona tiene 6 vecinos. Esta es la opción preferida si los datos tienen relaciones espaciales que se representan mejor con una cuadrícula hexagonal. Este es el caso de distribuciones de datos circulares o angulares.
- Número de iteraciones de entrenamiento (
N_ITERACIONES): En principio, tiempos de entrenamiento más largos conducen a errores más bajos y una mejor alineación de los pesos con los datos de entrada. Sin embargo, el rendimiento del modelo aumenta asintóticamente con el número de iteraciones. Por lo tanto, después de un cierto número de iteraciones, el aumento de rendimiento de las interacciones subsiguientes es solo marginal. Decidir el número correcto de iteraciones requiere algo de experimentación. En este tutorial, entrenamos el modelo durante 5000 iteraciones.
Para determinar la configuración adecuada de hiperparámetros, recomendamos experimentar con varias opciones en un subconjunto más pequeño de los datos.
Conclusión
Los mapas autoorganizados son una herramienta robusta para aprendizaje no supervisado. Se utilizan para la agrupación, reducción de dimensionalidad, detección de anomalías y visualización de datos. Dado que preservan las propiedades topológicas de datos de alta dimensión y los representan en una cuadrícula de menor dimensión, los SOMs facilitan la visualización e interpretación de conjuntos de datos complejos.
Este tutorial discutió los principios subyacentes de los SOMs y mostró cómo implementar un SOM utilizando la biblioteca MiniSom de Python. También demostró cómo analizar visualmente los resultados y explicó los importantes hiperparámetros utilizados para entrenar los SOMs y ajustar su rendimiento.
Source:
https://www.datacamp.com/tutorial/self-organizing-maps













